| Просмоторщики графики — это специальные компьютерные программы предназначенные для | ||||||||||||||||||||||||||
| ||||||||||||||||||||||||||
Диалоговое окно Предварительный просмотр графики
В этом диалоговом окне отображается предв.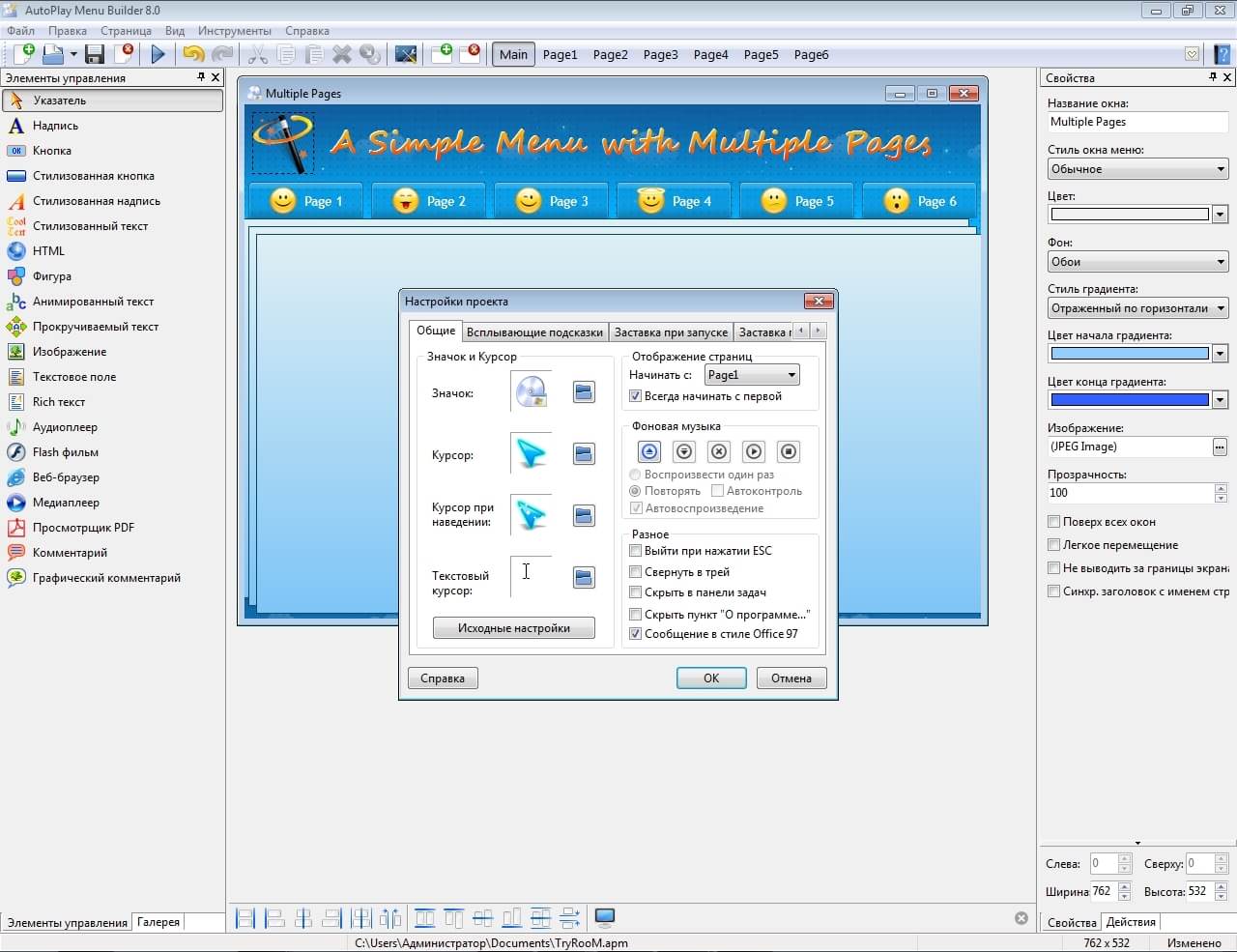 просмотр страниц, символов, графических файлов, макросов, форм и т. д., при этом текущий выделенный объект представлен в диалоговом окне выбора, если там установлен флажок Предв. просмотр. Это относится, например, к выбору макросов и форм и т. д.
просмотр страниц, символов, графических файлов, макросов, форм и т. д., при этом текущий выделенный объект представлен в диалоговом окне выбора, если там установлен флажок Предв. просмотр. Это относится, например, к выбору макросов и форм и т. д.
Обзор основных элементов диалогового окна:
Страница в предварительном просмотре выводится точно как так же, как и в графическом редакторе. Выводится вся информация, имеющаяся на странице, даже если эта информация находится за пределами . При отображении учитываются рамка и размер страницы.
- Если в диалоговом окне навигатора Страницы — <Имя проекта> выделена страница, эта страница выводится в режиме предварительного просмотра графики. Если выделено несколько страниц, на предварительный просмотр они выводятся друг под другом и рядом друг с другом.
- Если в навигаторе, в котором отображаются данные (например, Устройство — <Имя проекта>, Кабели — <Имя проекта> и т.
 д.), выделена одна из функций, то в предварительном просмотре графики будет отображена та страница, на которой находится эта функция. Позиция такой выделяется.
д.), выделена одна из функций, то в предварительном просмотре графики будет отображена та страница, на которой находится эта функция. Позиция такой выделяется. - Если в диалоговом окне Управление сообщениями — <Имя проекта> выделено какое-либо сообщение, то в предварительном просмотре графики будет отображена страница, к которой относится это сообщение, где будет выделена ошибочная функция.
- Если в диалоговом окне Список устройств выделена какая-либо запись, в графическом предварительном просмотре отображается символ или макрос, который используется для соответствующего изделия. Если добавить в список устройств новое , также выводится символ или макрос, использованный для этого изделия.
- Если в поле Выбор устройства выделено какое-либо изделие, в графическом предварительном просмотре выводится графический файл, который используется для соответствующего изделия.
- Если в диалоговом окне Генерировать схему соединений ПЛК выделена запись для файла макроса, то в графическом предварительном просмотре отображаются содержащиеся там макроса.

- Если в диалоговых окнах Выбрать макрос, Выбрать рамку, Выбрать форму … установлен флажок Предв. просмотр, в предварительном графическом просмотре отображается соответствующий объект.
- Если в навигаторе основных данных изделий выделено какое-либо изделие, которому в базе данных изделий на вкладке Шаблоны функций присвоены данные в поле Выбор устройства, то в предварительном просмотре графики будет отображено предварительное изображение этого устройства, и это изображение можно будет разместить в графическом редакторе при помощи перетаскивания мышью.
- Если в диалоговом окне навигатора Опции — <Имя проекта> выделен фрагмент, то в предварительном просмотре графики отображается его содержимое. Если выделено несколько фрагментов, они последовательно (друг под другом и рядом друг с другом) отображаются в предварительном просмотре.
См. также
Вывод предварительного отображения графики
Диалоговое окно Страницы — <Имя проекта>
Просмотр маршрутов для транспортировок (Библиотека SAP
Просмотр маршрутов для транспортировок
Использование
Просмотр одной
или более транспортировок возможен на карте дорог на закладке Географический обзор в интерактивном
планировании. Маршрут можно просмотреть как дорожный маршрут, т.е. точный
маршрут, подлежащий следованию, или как символический маршрут, т.е. прямое
сообщение между отдельными местоположениями. Возможен также просмотр описаний
маршрута со временем в пути и расстоянием поездки на основе дорожной
сети.
Маршрут можно просмотреть как дорожный маршрут, т.е. точный
маршрут, подлежащий следованию, или как символический маршрут, т.е. прямое
сообщение между отдельными местоположениями. Возможен также просмотр описаний
маршрута со временем в пути и расстоянием поездки на основе дорожной
сети.
Предпосылки
● Внешний сервер ГИС (ГИС = географическая информационная система) и IGS (сервер интернет-графики) установлены и соединены с SAP APO.
● Активирован просмотр карты для пользователя в пользовательской настройке компонента Планирование транспортировки
● Созданы географические координаты для местоположений.
Функции
Просмотр маршрутов
Маршруты
представлены в виде цветных линий. Начальные и целевые местоположения и
промежуточные остановки для лучшего обзора снабжены описанием в дополнение к
графическому просмотру.
Возможен просмотр маршрутов для транспортировок и для заказов и ресурсов, которым присвоены транспортировки. При выборе заказов обратите внимание на то, что в случае транспортировки с несколькими заказами, система представляет маршрут только до целевого местоположения выбранных заказов.
Если во время просмотра происходит переключение закладки и выбор другого объекта в строке кнопок (левая часть экрана), обновление просмотра карты дорог произойдет при повторном выборе закладки Географический обзор.
Просмотр описания маршрута, времени в пути и расстояния
Для просмотра подробного описания маршрута для этой транспортировки следует выбрать Маршрут.
Если выбрать
стрелку справа от Выбранной транспортировки и затем выбрать требуемую транспортировку, время в пути и
расстояние отображаются на основе дорожной сети для этой транспортировки.
Расстояние отображается в определенной пользователем единице (например, миля,
км).
Для просмотра времени в пути и расстояния для всех отображенных транспортировок следует выбрать Обзор.
Удаление транспортировок из просмотра
Для удаления выбранных транспортировок из карты дорог следует выбрать Удалить.
Если справа от Выбранной транспортировки не выбрана транспортировка, то все отображенные на карте дорог транспортировки будут удалены при выборе функции
Если на карте отображено несколько транспортировок, а выбрана только одна транспортировка справа от Выбранной транспортировки, можно отменить выбор этой выбранной транспортировки путем выбора стрелки справа от Выбранной транспортировки и затем пустой записи.
Изменение формата карты дорог
Карту дорог можно центрировать, увеличивать и уменьшать. Также возможно перемещение фрагмента карты и возврат к общему ракурсу в любое время.
Действия
. ..
..
1. На экране SAP Easy Access выберите Advanced Planner and Optimizer ® Планирование транспортировки/планирование транспортных средств ® Планирование ® Интерактивное планирование транспортных средств
.2. Выберите профиль оптимизации и с текстом всплывающей подсказки Выполнить.
3. Выберите закладку Географический обзор.
4. Выберите одну или более транспортировок в строке кнопок и затем Просмотр. Появится соответствующий раздел карты дорог и маршрутов транспортировки.
Бесплатная программа для просмотра изображений
Бесплатная программа для просмотра изображений с множеством дополнительных функций, включая конвертор, редактор, браузер, режим слайд-шоу (более 150 вариантов переходов) и пакетное преобразование.
Возможности просмотрщика
EXIF (англ. Exchangeable Image File Format) — метаданные, дополнительная информация, «вшитая» в файл изображения.Слайд-шоу (англ. slideshow) — демонстрация серии неподвижных картинок.
- Быстрый и удобный просмотр всевозможных картинок.
- Снятие скриншотов любой части экрана.
- Поддержка популярных форматов: JPEG, JPEG 2000, BMP, GIF, PNG, TIFF, PCX, WMF, PSD (родной формат фотошопа), RAW (формат для хранения исходников профессиональных фотографий), ICO (иконки), TGA, CRW (Canon), CR2 (Canon), NEF (Nikon), ORF (Olympus), RAF (Fuji), DNG (Adobe), MRW (Minolta), PEF (Pentax), SRF (Sony), ARW (Sony) и RW2 (Panasonic).
- Удобный и понятный интерфейс (есть сменные «шкурки»).
- Редактирование метаданных (EXIF).
- Экспорт изображений в формат PDF.
- Не содержит рекламных и шпионских модулей.
- Встроенный редактор изображений (в том числе, позволяет убирать эффект красных глаз).
- Пакетная обработка и конвертация картинок.

- Создание слайд-шоу и много чего ещё.
В общем и целом, это отличная замена платному ACDSee.
Установка FastStone Image Viewer
Скачиваем и достаём из архива запускаемый файл программы и… правильно, запускаем его.
Поочерёдно открываются стандартные окна установки программ, в которых нажимаем: «Далее», «Принимаю», «Установить» и «Готово». То есть, всё как обычно, никаких сюрпризов.
Интерфейс программы
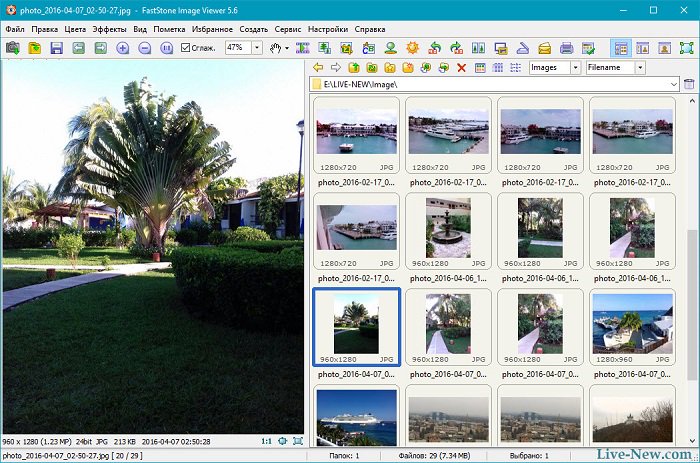
Структура главного окна программы напоминает обычный Проводник. Слева — браузер и предпросмотр выбранной картинки, а справа — галерея картинок, находящихся в данной папке.
Здесь можно сразу отметить, что программа запоминает последнюю открытую картинку и путь к ней. Это очень удобно, если нужно продолжить работать с текущей папкой после перезагрузки компьютера.
Настройки
Есть некоторые первичные настройки, которые стоит сделать сразу, для удобства дальнейшего пользования программой. Чтобы попасть в окно настроек, нажимаем клавишу F12 или заходим в меню «Настройки» — «Настройки программы».
И первое, что мы делаем, это во вкладке «Просмотр» включаем по умолчанию «Оконный режим» просмотра картинок.
Данная настройка позволит нам всегда иметь перед глазами панель инструментов.
Если этого не сделать, то просмотр будет производиться в полноэкранном режиме на чёрном фоне. А для доступа к панели (например, чтобы распечатать картинку) придётся каждый раз подносить курсор мыши к краю экрана.
Ассоциирование — связывание. Файл будет открываться в этой программе по умолчанию.Второе, это ассоциирование графических файлов с FastStone Image Viewer. Для этого переходим на вкладку «Ассоциации» и отмечаем галочками все форматы изображений, которые хотим просматривать этой программой.
Если Вы не профессиональный дизайнер, то можете просто нажать кнопку «Выбрать все» и подтвердить «Ок».
Собственно, это все настройки, которые я использую сам, когда ставлю эту программу на новый компьютер. Остальные можно оставить по умолчанию — они уже достаточно удобны.
Здесь мы упомянем их лишь для полноты картины:
Превьюшка (англ. preview) — предпросмотр. Уменьшенная версия картинки или кадр видеоролика, дающие представление о полной версии, но занимающие меньше места на экране.RAW (англ. сырой, необработанный) — формат для хранения исходных данных с матрицы фотоаппарата.
- Просмотр — параметры окна просмотра, позволяющие подогнать его под любой вкус.
- Миниатюры — настройка внешнего вида превьюшек, начиная от размера и заканчивая скруглением рамки.
- Лупа — позволяет выбрать увеличение, размер и фон лупы, которая включается в окне просмотра при нажатии и удержании левой кнопки мыши.
- JPEG — качество сохранения в формат JPG.
- Raw — параметры просмотра RAW-файлов.
- Второй монитор — при наличии дополнительного монитора можно разнести полный просмотр и миниатюры на разные экраны.
- CMS — включение/отключение системы управления цветом.
- Избранное — можно добавить в программу избранные папки, к которым приходится чаще всего обращаться.

- Программы — подключение внешних программ для обработки файлов.
- Музыка — выбор музыки для проигрывания слайд-шоу.
Полный просмотр изображения
Для включения полного просмотра достаточно дважды кликнуть левой кнопкой мыши на превью нужной картинки. Откроется вот такое окно:
Здесь мы можем в подробностях разглядеть нашу картинку. Если она всё ещё мелковата, используем Лупу (нажимаем и удерживаем левую кнопку мыши).
С помощью меню и инструментов в верхней части экрана можно произвести различные операции над изображением, например:
Водяной знак — полупрозрачная надпись или картинка поверх основного изображения.Ретушь — устранение шумов и ненужных деталей на фотографии.
- изменить размер;
- обрезать края;
- нарисовать что-нибудь поверх в простом встроенном редакторе;
- добавить водяной знак;
- сделать ретушь фотографии;
- скорректировать цвета;
- повернуть на прямой угол или отразить по вертикали/горизонтали;
- применить разнообразные фильтры и рамки;
- распечатать;
- отправить по электронной почте;
- и многое другое.

Вернуться в режим браузера можно всё тем же двойным кликом мышки по картинке.
Полноэкранный просмотр
Иногда удобнее просматривать фотографии на простом чёрном фоне без лишних элементов. В таком случае поможет полноэкранный просмотр.
Он включается с помощью соответствующей иконки в панели управления, либо просто кнопкой F на клавиатуре.
На этом скриншоте видно, что если подвести курсор мыши к одной из четырёх сторон экрана, появляются разные менюшки, позволяющие работать с картинкой даже в полноэкранном режиме.
А теперь, давайте рассмотрим некоторые, наиболее интересные возможности программы.
Удаление эффекта красных глаз
Красные глаза — эффект отражения вспышки фотоаппарата от капилляров глазного дна.Чтобы скорректировать этот неприятный эффект, открываем нужную фотографию, заходим в меню «Правка» — «Уменьшение эффекта красных глаз».
Откроется вот такое незамысловатое окошко, в котором нужно лишь по очереди выделить красные зрачки и нажать кнопку «Удалить красноту».
Если фокус не удался, можно вернуть как было кнопкой «Отменить».
Коррекция цвета
Фотографии порой получаются настолько же важные, насколько и неудачные в плане осветлённости и баланса цветов.
Чтобы спасти ценный кадр, можно воспользоваться функцией коррекции цвета в FastStone Image Viewer.
Для этого нажимаем кнопку с оранжевым солнышком в панели инструментов или нажимаем Ctrl + T. В меню это пункт «Цвета» — «Коррекция освещённости».
В открывшемся окне можно подрегулировать свет, тень, контрастность и насыщенность фотографии.
Выбор настроек здесь не велик, но они достаточно просты, что поможет начинающим не запутаться.
Для продвинутых фотографов в меню «Цвета» есть дополнительные наборы профессиональных инструментов:
- Коррекция цветов.
- Коррекция уровней.
- Коррекция кривых.
- Резкость / размытие.
- Шумоподавление.
- Уменьшение количества цветов.
- Подсчёт цветов — выдаёт суммарное количество разных цветов на фото.

- Оттенки серого — эффект «лазерной ксерокопии».
- Сепия — коричневый цвет «под старые фотографии».
- Негатив — инверсия цветов.
С их помощью можно достаточно гибко обрабатывать сделанные снимки, получая приличный результат с минимумом усилий.
Эффекты
Несколько любопытных инструментов спрятано в меню «Эффекты»:
- Рамки — добавляет простую прямоугольную рамку, для которой можно настроить цвет, тень и т.д.
- Трафареты — довольно солидный набор фигурных рамок типа «сердечко», «листик» и т.п.
- Водяной знак — добавляет на изображение любую другую картинку (например, текст на прозрачном фоне), для которой можно задать тень и прозрачность.
- Рельеф — эффект барельефа, где тёмные цвета становятся углублениями, а светлые — выпуклостями.
- Эскиз — имитация карандашного наброска. Результат оставляет желать лучшего 🙂
- Линза — создаёт эффект выпуклости в отдельно взятом месте картинки.
- Размытие — размывает выделенную область (для случаев, когда нужно частично скрыть какую-то конфеденциальную информацию).

- Картина маслом — довольно натуральный фильтр «под масляную краску» в стиле художника Пластова.
Рисование в FastStone Image Viewer
Следующая интересная возможность — рисование прямо поверх картинки. По правде сказать, именно таким образом я добавляю пояснительные надписи на скриншоты в наших статьях.
Давайте рассмотрим это окно поподробнее. Нажимаем кнопку «Рисование» на панели инструментов или заходим в меню «Правка» — «Рисование».
Полноценных инструментов художника здесь нет, но для создания пометок программа подходит отлично.
Здесь есть важный момент. Все добавленные объекты можно редактировать лишь до тех пор, пока не нажата кнопка «Ок». После этого они уже будут являться неотъемлимой частью исходного изображения.
Меню «Создать»
В данном пункте меню собраны следующие функции:
EXE — формат самостоятельных приложений.- Создать файл со слайд-шоу (в формате EXE).
- Создать многостраничный файл (в формате PDF, TIFF или Анимированный GIF).

- Разделить многостраничный файл.
- Создать ленту изображений (несколько картинок в ряд).
- Создать индекс-лист (большая графическая схема с превьюшками выбранных изображений).
Меню «Сервис»
В последнем наборе инструментов нам доступны следующие полезные возможности:
- Преобразовать выбранные.
- Переименовать выбранные.
- Пакетное преобразование/переименование.
- Изменить дату/время выбранных.
- Поворот JPEG без потерь.
- Удалить метаданные JPEG.
- Сравнить изображения.
- Обои.
Первые три пункта по сути являются разными вкладками одного и того же окна. Они позволяют довольно гибко изменять имена или параметры не одной а сразу множества фотографий.
К примеру, можно уменьшить линейные размеры выбранных фотографий, сжать их, добавить водяной знак и сразу же, автоматически переименовать по шаблону, чтобы не испортить оригиналы. Неоценимый инструмент для свадебных фотографов 🙂
Последний пункт «Обои» позволяет сделать выбранный рисунок фоном на Рабочем столе Windows.
Сравнение изображений
Пожалуй, напоследок рассмотрим ещё один любопытный инструмент: Сравнение 2-х или 4-х рисунков на одном экране.
Для этого нужно выделить 2 или 4 превьюшки в браузере и нажать кнопку «Сравнить выбранные изображения».
На выходе получим вот такую картину:
Здесь можно посмотреть не только сами рисунки, но и их гистограмму, а также метаданные (EXIF), если они есть.
Выводы
FastStone Image Viewer — настоящий шедевр в своём роде. Он идеально подходит для просмотра изображений, потребляет мало ресурсов, весит меньше 6 МБ, гибко настраивается и очень удобен в использовании.
Это была одна из первых программ, появившихся на нашем сайте и мы до сих пор ею активно пользуемся, как для просмотра, так и для несложного редактирования картинок. Поэтому она заслуженно получила золотую медаль «Выбор BestFREE.ru».
P.S. Разрешается свободно копировать и цитировать данную статью при условии указания открытой активной ссылки на источник и сохранения авторства Руслана Богданова.
P.P.S. Если простого просмотра Вам недостаточно, попробуйте несложный редактор фотографий Paint.NET https://www.bestfree.ru/soft/graph/paint.php.
Топ-8 GUI-клиентов для PostgreSQL в 2021 году / Хабр
Что такое графический интерфейс PostgreSQL? Зачем он вам нужен? Как он может помочь управлять базой данных PostgreSQL? Узнайте о лучшем программном обеспечении Postgre GUI, которое можно попробовать в 2021 году.
PostgreSQL — это передовая объектно-реляционная система управления базами данных с открытым исходным кодом. В основном, она используется на предприятиях и поддерживает SQL и JSON запросы.
По данным Stack Overflow, PostgreSQL является второй по использованию базой данных после MySQL в 2021 году. Более 40% из 70 000+ респондентов предпочитают Postgres по сравнению с SQLite, MongoDB, Redis и другими базами данных.
Для пользователя Postgres существует два способа администрирования базы данных:
Писать запросы через командную строку (CLI) (не всем это нравится).

Использовать графический интерфейс пользователя Postgres (GUI), созданный с помощью одного из инструментов управления PostgreSQL.
Второй вариант гораздо удобнее, чем CLI. Кроме того, он позволяет повысить производительность труда. Сейчас мы познакомим вас с Postgres GUI и наиболее часто используемыми инструментами PostgreSQL GUI.
Что такое PostgreSQL GUI?
PostgreSQL GUI — это инструмент управления базами данных PostgreSQL. Он позволяет вам или любому пользователю базы данных запрашивать, визуализировать, манипулировать и анализировать данные Postgres. Вы также можете получить доступ к серверам баз данных и осуществлять навигацию по ним с помощью Postgres GUI.
Основными причинами, по которым пользователи предпочитают Postgres GUI, а не CLI, являются:
Длительная кривая обучения и сложный процесс адаптации в работе с CLI.
Не очень приятный в использовании интерфейс CLI.
Недостаток информации, которую предоставляет консоль за один раз.

Сложности с просмотром и мониторингом баз данных через консоль.
Использование Postgres GUI, в свою очередь, предлагает вам следующие преимущества:
Ярлыки, с помощью которых вы можете быстрее и проще осуществлять доступ к необходимым данным.
Широкие возможности визуализации данных.
Удаленный доступ к серверу базы данных.
Легкий доступ к операционной системе.
Лучшее программное обеспечение PostgreSQL GUI
Возможно, для кого-то будет неожиданностью тот факт, что ориентированный на Postgres pgAdmin — не единственный инструмент Postgres GUI, доступный сегодня.
Во-первых, есть UI Bakery, низкоуровневый конструктор внутренних инструментов. Изначально он не был создан для управления Postgres. Однако, с его помощью вы сможете подключить несколько источников данных (базы данных, сторонние приложения, REST API) в одном пользовательском интерфейсе. UI Bakery также предоставляет широкие возможности визуализации данных, позволяя отображать содержимое PostgreSQL, MongoDB, MySQL, Microsoft SQL, Redis и других баз данных в нужном виде.
Поскольку UI Bakery основан на веб-технологиях, не требуется тратить время на его установку и настройку. Можно создать графический интерфейс для своей базы данных Postgres, используя ряд готовых компонентов, таких как таблицы, диаграммы, графики, карты, кнопки, выпадающие окна и т.д. Этот процесс занимает минуты или часы вместо дней и недель ручного кодирования.
Подход к управлению базами данных с использованием низкоуровневого кода гораздо экономичнее по времени и более гибок, чем использование традиционных графических инструментов. Тем не менее, давайте теперь рассмотрим и другие инструменты управления GUI.
1. pgAdmin
Кроссплатформенный графический интерфейс PostgreSQL с открытым исходным кодом.
Преимущества:
Совместимость с Linux, Windows, macOS.
Позволяет работать с несколькими серверами одновременно.
Экспорт файлов в формате CSV.
Планирование запросов.

Возможность отслеживания сессий, блокировок базы данных через панель мониторинга.
Ярлыки в редакторе SQL для более удобной работы.
Встроенный отладчик процедурного языка, который облегчает процесс доводки кода.
Тщательная документация и активное сообщество.
Недостатки:
Медленный и недостаточно интуитивно понятный пользовательский интерфейс по сравнению с платными конкурентами.
Слишком тяжелый.
Сложность в освоении.
Для одновременной работы с несколькими базами данных требуются продвинутые навыки.
2. DBeaver
Инструмент управления PostgreSQL с открытым исходным кодом, поддерживающий несколько баз данных.
Преимущества:
Кросс-платформенность.
Поддержка более 80 баз данных.
Визуальный конструктор, позволяющий добавлять запросы без навыков работы с SQL.

Возможность создания нескольких представлений данных.
Импорт/экспорт данных в форматах CSV, HTML, XML, JSON, XLS, XLSX.
Повышенная безопасность данных.
Полнотекстовый поиск данных и возможность отображения результатов поиска в виде таблиц/представлений.
Доступен бесплатный тарифный план.
Недостатки:
Медленная производительность по сравнению с конкурентами.
Слишком частые обновления, что раздражает.
После некоторого времени бездействия DBeaver отключается от вашей базы данных. Вам нужно заново запускать приложение.
3. Navicat
Достаточно интуитивно понятный графический инструмент для управления базами данных Postgres. Не имеет открытого исходного кода.
Преимущества:
Очень простая и быстрая установка.
Поддержка Windows, Linux, macOS, iOS.
Удобный и быстрый визуальный конструктор SQL.

Функция автодополнения кода.
Инструмент моделирования данных: работа с объектами базы данных, схемами проектирования.
Планировщик заданий: запуск заданий, получение уведомлений о завершении заданий.
Встроенная функция совместной работы в команде.
Синхронизация источников данных.
Импорт/экспорт данных в Excel, Access, CSV и других форматах.
Защита данных с помощью SSH и SSL.
Использование облачных сервисов Amazon, Google и других.
Недостатки:
Низкая производительность GUI.
Довольно высокая цена по сравнению с конкурентами.
Одна лицензия ограничена одной платформой (для PostgreSQL и MySQL нужны две отдельные лицензии).
Большое количество дополнительных функций, требующих времени для обучения работе с ними.
Неудобство: необходимо обновлять приложение при добавлении строк.
4. DataGrip
DataGrip
Продвинутая IDE с поддержкой нескольких баз данных, созданная компанией JetBrains.
Преимущества:
Кроссплатформенность (поддержка Windows, macOS, Linux).
Простая навигация по схеме.
Настраиваемый пользовательский интерфейс с консолью запросов для обеспечения безопасности рабочего процесса.
Быстрое обнаружение ошибок.
Встроенная система контроля версий.
Поддержка MySQL, SQLite, MariaDB, Cassandra и других баз данных.
Наглядные отчеты с возможностью интеграции их с диаграммами и графиками.
Отличная функция автодополнения, предлагающая подходящие варианты завершения кода.
Недостатки:
Достаточно дорогой.
Потребляет много оперативной памяти.
Сложный процесс отладки ошибок.
Длительная кривая обучения как для DataGrip, так и для JetBrains.
Не предназначен для использования в качестве облачного веб-приложения.

Не подходит для управления несколькими базами данных одновременно.
5. HeidiSQL
Инструмент GUI для Postgres (и не только) с открытым исходным кодом. Пока поддерживается только Windows.
Преимущества:
Простая установка, достаточно легковесный по сравнению с конкурентами.
Поддержка PostgreSQL, MySQL, Microsoft SQL Server, MariaDB.
Возможность подключения и управления несколькими серверами баз данных в одном окне.
Прямой экспорт SQL из одной базы данных или сервера в другую.
Массовый просмотр и редактирование таблиц с помощью простой в использовании грид-системы.
Функции автодополнения кода и подсветки синтаксиса.
Активное сообщество поддержки, регулярно совершенствующее этот GUI инструмент.
Экспорт таблиц и данных в Excel, HTML, JSON, PHP.
100% зашифрованное соединение.
Недостатки:
Не является кроссплатформенным (поддерживается только Windows).

Часто возникают проблемы со стабильностью.
Нет отладчика процедурного языка для простой доводки кода.
6. TablePlus
Нативное программное обеспечение с графическим интерфейсом для управления базами данных SQL и NoSQL. С закрытым исходным кодом.
Преимущества:
Высокая производительность и скорость работы (по отзывам пользователей).
Хорошо кастомизируемый пользовательский интерфейс: вам совсем не нужно переходить на Mojave.
Поддержка подсветки синтаксиса.
Доступны быстрые клавиши для экономии времени и повышения эффективности.
Высокий уровень безопасности данных благодаря сквозному шифрованию соединения клиент-сервер.
Недостатки:
Часто возникают проблемы с UX при работе с другими базами данных, кроме PostgreSQL.
Недешево. Бесплатная пробная версия предлагает крайне ограниченную функциональность.

Поддержка клиентов оставляет желать лучшего.
7. OmniDB
Простой GUI инструмент управления для PostgreSQL с открытым исходным кодом .
Преимущества:
Кроссплатформенность (поддержка Windows, Linux, macOS).
Поддержка PostgreSQL, Oracle, MySQL, MariaDB.
Очень отзывчивый и легковесный по сравнению с некоторыми альтернативами.
Автозаполнение SQL.
Функция подсветки синтаксиса.
Возможность создания настраиваемых диаграмм для отображения соответствующих показателей базы данных.
Встроенная функция отладки.
Недостатки:
Не самый подходящий вариант, если вы работаете с несколькими базами данных одновременно.
Отсутствие поддержки и обучающей документации.
Резюмируя: UI Bakery — неочевидный, но мощный конкурент.
Когда вы выбираете программное обеспечение с графическим интерфейсом, при принятии окончательного решения учитывайте несколько аспектов:
Размер команды.

Ваша операционная система.
Тип базы данных.
Количество баз данных, с которыми вы планируете работать.
DBeaver, DataGrp и HeidiSQL больше подходят для одного человека, работающего с одной базой данных. Navicat — выбор для команды благодаря возможности совместной работы с GUI. Почти все перечисленные инструменты являются кроссплатформенными, за исключением HeidiSQL, который поддерживается только Windows. PgAdmin, ориентированный на PostgreSQL, является довольно мощным GUI-инструментом для нее.
Но есть UI Bakery, визуальный конструктор внутренних инструментов. Эта низкоуровневая платформа разработки отлично подходит, если вам нужно объединить несколько различных источников данных — будь то базы данных, сторонние инструменты или API. Вам не обязательно оставаться в рамках только одной экосистемы. Можно также использовать готовые компоненты и шаблоны пользовательского интерфейса, чтобы не начинать с нуля и сэкономить свое время.
Если вы не уверены, подходит ли конструктор внутренних инструментов под ваши конкретные нужды, попробуйте низкоуровневый подход. На весь процесс разработки инструмента GUI уйдут часы или даже минуты, в зависимости от вашего опыта разработки.
Похоже, что pgAdmin и другое классическое ПО с GUI теряют популярность. Низкоуровневый подход к управлению базами данных Postgres (и других) позволяет получить гораздо лучшие результаты за меньшее время.
Материал подготовлен в рамках курса «Базы данных».
Всех желающих приглашаем на demo-занятие «Миграция данных». На занятии разберем виды миграции и основные проблемы, возникающие при миграции данных. На примере PostgreSQL посмотрим, как можно легко и быстро перейти с одной версии СУБД на другую. >> РЕГИСТРАЦИЯ
На художественно-графическом факультете ДГПУ прошел экзаменационный просмотр
2 июня на художественно-графическом факультете Даггоспедуниверситета прошёл экзаменационный просмотр по специальностям «живопись», «графика» и «скульптура».
Работы студентов оценивала комиссия, в которую вошли заведующий кафедрой живописи, член-корреспондент Российской академии художеств, профессор Абдулзагир Мусаев, декан художественно-графического факультета Хабиб Омаров, народный художник Дагестана Юсуп Ханмагомедов и другие преподаватели факультета. Гостями просмотра стали и.о. ректора Даггоспедуниверситета Нариман Асваров, председатель Союза художников Дагестана Курбанали Магомедов, член Ассоциации искусствоведов Екатерина Дидковская и др.
Студенты-скульпторы представили на просмотр этюды мужской головы и женской фигуры, эскизы заданий по композиции. Работы были отобраны для формовки и последующей отливке в гипсе.
Студенты мастерской графики представили свои работы по живописи и рисунку, а также печатную графику – офорты, ксилографии и линогравюры. Заданием этого семестра также было создание книжных иллюстраций для детской и взрослой аудитории и книжного знака – экслибриса. Работы студентов-графиков получили отличные оценки и были рекомендованы для отбора в методический фонд факультета.
Учащиеся мастерской живописи выполнили за семестр несколько учебных постановок – портреты натурщиков на светлом и темном фоне, натюрморт и фигура на сложном фоне. По предмету «Композиция» были представлены темы «Архитектура», «Профессии». Комиссия дала высокие оценки представленным работам и отметила отличный уровень выполнения студентами поставленных задач.
По окончании просмотра члены комиссии и гости провели беседу со студентами, прокомментировав работу каждой мастерской, пожелали дальнейших успехов в освоении профессии. В завершение наиболее отличившиеся студенты были награждены дипломами Союза художников Дагестана.
Посетить кафедру живописи и мастерские, чтобы увидеть работы студентов, могут все желающие с 3 по 11 июня.
Иллюстрированный самоучитель по Linux › KDE › Утилиты KDE. Просмотр текста и графики. [страница — 167] | Самоучители по операционным системам
Утилиты KDE. Просмотр текста и графики.
В комплект KDE входит ряд стандартных утилит для просмотри текста и графики, управления проектами и администрирования системы Linux. Рассмотрим наиболее популярные из них.
Рассмотрим наиболее популярные из них.
В KDE есть текстовый редактор, программа просмотра изображений и графический редактор (аналогичный программе paintbrush). Запуск этих программ несложен. Стандартный текстовый редактор KEdit запускается выбором в главном меню элемента Editors (Редакторы), а в нем – команды Text Editor (Текстовый редактор). Для доступна к более мощному редактору текстов KWrite следует выбрать в главном меню элемент Editors (Редакторы), а в нем – команду Advanced Editor (Усовершенствованный редактор).
Аналогичным образом через подменю Graphics (Изображения) возможен доступ к ряду программ просмотра изображений, включая KView (подобная Image Viewer), KDE Pixmap2Bitmap и Xpaint. Можно также обратиться к The GIMP как к инструменту редактирования изображения, воспользовавшись тем же самым подменю Graphics.
Щелчок правой кнопкой мыши на соответствующем файле в окне диспетчера файлов Konqueror позволяет открыть файл, используя проходящее приложение. Например, после щелчка правой кнопкой мыши на тестовом файле и выбора в контекстном меню команды Open With (Открыть с помощью) можно указать необходимое для открытия файла приложение (рис. 11.18).
Например, после щелчка правой кнопкой мыши на тестовом файле и выбора в контекстном меню команды Open With (Открыть с помощью) можно указать необходимое для открытия файла приложение (рис. 11.18).
Рис. 11.18. Использование диспетчера файлов Konqueror для открытия текстового файла с выбором приложения
Если необходима отсутствующая в подменю программа, щелкните на Other (Другие). В результате откроется окно диалога Open With (Открыть с помощью), позволяющее выбрать приложение из главного меню KDE.
Легенда графического представления
Эта легенда относится к программе просмотра последовательности NCBI. NCBI Genome Workbench использует аналогичные графические представления.
- Визуализация общих функций
- Особенности модели гена
- Особенности размещения клонов
- Характеристики SNP
- Варианты конструкции
- Сегментарные дублирования
- Раскладки
- Дорожка последовательности
- Карта сегмента
- Шесть кадровых переводов
- Размещение этикетки
- Отрисовка гистограммы или графика
1.
 Отрисовка общих функций
Отрисовка общих функцийВ этом разделе описывается отрисовка, используемая для функций генов, моделей РНК и белков, регуляторных сайтов и большинства других типов функций. Эти функции часто происходят из файлов, отформатированных как BED, bigBED, GFF3, GTF, ASN.1 и аналогичные форматы.
Специализированный рендеринг для SNP, структурных вариантов и сегментных дубликатов описан в следующих разделах этой документации.
1.1 Код цвета элемента
Белые стрелки внутри объектов указывают направление объекта относительно последовательности верхнего уровня.Серые стрелки в интронах (например, в характеристиках РНК и кодирующей области) указывают направление сплайсинга.
1.2 Параметры отображения функций
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks .
Пользователи могут установить разрешение дисплея с помощью раскрывающегося меню Параметры рендеринга . По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
Параметр , связывающий параметры , обеспечивает различные виды отображения для дорожек, содержащих родительские и дочерние функции; т.е. «Показать все»; «Показать родителя, объединить потомков»; «Показывать детей, а не родителей»; «Показывать родителей, а не детей»; «Наложите детей на родителя». Изменение этих параметров может привести к отображению большего или меньшего количества функций на дорожке, в зависимости от базового источника данных на дорожке.
1.2.1 Отображение функции интрона
Дорожки интрона отображают сплайсинговые соединения экзон-экзон на основе анализа данных RNA-seq. Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Intron содержат дополнительные параметры отображения дорожек, в том числе возможность сортировать элементы стыка сращивания по количеству считываний сращивания или по направлению сращивания (прядям).Пользователи также могут упростить отображение этой дорожки, фильтруя данные по количеству совмещенных считываний.
1.3 Специальные стили визуализации
1.3.1 Псевдогеновые признаки
| Настройки дисплея | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Показать все | Полосы в зеленой полосе гена | |
| Без генной полосы | Зеленая полоса фона | |
| Показать на одной строке со структурой экзона, без генной полосы | Зеленая полоса фона |
1.
 3.2 Элементы с несовершенным соответствием сборке
3.2 Элементы с несовершенным соответствием сборке| Пример | Визуальный эффект | Визуальные примеры |
|---|---|---|
| РНК или CDS с несоответствием последовательности относительно сборки | Затененный фон |
1.3.3 Элементы с частичным расположением аннотаций (например, из-за ошибок сборки или зазоров)
| Примеры | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Частичный 5 ‘конец | Черные стрелки («<<» или «>>») на конце 5 ‘ | |
| Частичный 3 ‘конец | Черные стрелки («<<" или ">>») на конце 3 ‘ | |
| Внутренние неточные концы | Черные стрелки («<<» и «>>») на внутренних соединениях | |
| Частичные 5- и 3-дюймовые концы | Черные стрелки («<<» и «>>») на обоих концах |
1.
 3.4 Элемент, помеченный как частичный
3.4 Элемент, помеченный как частичный| Примеры случаев | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Элемент, отмеченный как частичный на концах 5 ’и 3’ (белые стрелки, «<<» и «>>»), и элемент с частичным концом 3 ’(черные стрелки« >> ») | Белые стрелки («<<» и «>>») на обоих концах или черные стрелки («>>») на одном конце. |
1.3.5 Ограничение возможностей сайта
Сайты узнавания рестрикционного фермента помечены треугольными метками, указывающими сайты разрезов.Треугольные метки отсутствуют при высоких уровнях масштабирования. Когда элемент выбирается щелчком мыши, расположение участков разреза обозначается красными линиями волос.
| Посмотреть | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Сайт узнавания рестрикционного фермента | Треугольники указывают места разрезов | |
| Место ограничения (функция выбрана) | Треугольники указывают место разреза; линиями волос отмечаются границы пространственных объектов и участки выреза | |
| Место ограничения (функция выбрана, уменьшено) | Без треугольников; линиями волос отмечаются границы пространственных объектов и участки выреза |
1.
 4 украшения
4 украшения| Стиль декора | Визуальный эффект | Визуальные примеры |
|---|---|---|
| По умолчанию | Сплошные столбцы для интервалов признаков или экзонов и сплошные линии для интронов | |
| Стрелки | Стрелки на обоих концах, показывающие нить, и заштрихованные полосы для интронов | |
| Квадратный анкер | Квадрат для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Круглый анкер | Круг для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Необычный | Круг для начала только мРНК, квадрат для начала других признаков, кроме гена и CDS, стрелка для остановки функции, заштрихованные столбцы для интронов мРНК и кривые линии для интронов CDS |
2.
 Особенности модели гена
Особенности модели генаХарактеристики генной модели состоят из нескольких возможных компонентов: генная панель, РНК / мРНК, CDS и характеристики экзона.
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks . Для треков аннотации генов NCBI или Ensembl могут быть доступны несколько вариантов. Параметр Hide non-coding скроет некодирующие транскрипты или некодирующие гены или псевдогены, чтобы отображались только варианты транскриптов, кодирующие белок. Hide model transcripts Функция скрывает модели стенограммы, предсказанные с помощью алгоритма Gnomon NCBI.
У вас также могут быть опции для отображения только вариантов, которые являются частью проекта CCDS или были обозначены как вариант RefSeq или MANE Select.
2.1 Визуализация генной модели
Настройки отображения трека включают несколько опций рендеринга, как описано ниже.
Когда все функции развернуты, столбцы генов окрашиваются в зеленый цвет, некодирующие РНК и элементы мРНК окрашиваются в фиолетовый цвет, а элементы CDS — в красный цвет.
При слиянии пар транскрипта и CDS кодирующие экзоны отображаются темно-зеленым цветом, а некодирующие области (т. Е. 5 ‘и 3’ UTR) отображаются более светлым оттенком зеленого, а некодирующие РНК остаются пурпурными (см. Пример «Объединить транскрипт и пары CDS, без генной полосы»).
Когда все транскрипты объединены и объединенные функции включают части экзонов, которые присутствуют в некоторых транскриптах, но отсутствуют в других транскриптах, интенсивность зеленой окраски пропорциональна количеству вариантов транскриптов, которые включают конкретную область экзона (см. «Объединить все транскрипты и CDS, без генной полосы », пример ниже).
Чтобы показать признаки, аннотированные на РНК или CDS, установите флажок Показать аннотированные признаки белка .
2.2 Специальная визуализация расхождений между аннотированными последовательностями RefSeq и геномными последовательностями
Визуализация несовпадений
В случае несинонимичных несовпадений, несоответствие выделяется комбинацией красного текста для кодона, отображения альтернативных оснований в мРНК и отображения альтернативных аминокислотных остатков в белковой характеристике.
В случае синонимичных несоответствий несоответствие выделяется комбинацией красного текста для кодона и отображения альтернативных оснований в мРНК.
Обработка вставок и удалений
Делеции отображаются в виде штрихов в соответствующих характеристиках мРНК и белков и отсутствуют какие-либо кодоны или аминокислоты.
Вставки отображаются в виде пары перевернутых вертикальных треугольников в мРНК и характеристиках белка, и показаны соответствующие кодоны и аминокислоты; кодоны окрашены в красный цвет.
2.3 Функциональная линейка
Когда все признаки развернуты в дорожке генной модели, при выборе объекта РНК или CDS, щелкнув по нему, появится линейка признаков, указывающая локальные координаты признака.
Когда выбрано несколько элементов, появляются линии волос, указывающие на различия в структуре разных элементов.
2.4 Подсказка по функциям
При наведении курсора на объект появляется всплывающая подсказка с дополнительной информацией об этом объекте.Всплывающая подсказка для функций генов, РНК и CDS сообщает о длине элемента, размахе сборки и координатах позиций, в которых всплывающая подсказка была активирована. Также включены ссылки на другие NCBI или внешние ресурсы, относящиеся к гену или функции, если таковые имеются. Также есть варианты для загрузки функции в форматах FASTA или GenBank, отправки ее в BLAST или открытия в ее собственном экземпляре графического средства просмотра последовательности. Более подробная информация о всплывающих подсказках для генов доступна здесь.
3. Особенности размещения клонов
3.1 Сообщаемые атрибуты
Графические изображения для всех элементов размещения клонов передают следующие атрибуты:
- Согласованность
- Уникальность
- Клонирование конца уверенности
- направленность и
- Подтверждающие доказательства
3.2 Визуальные примеры для переданного атрибута
3.3 Примеры визуализации для различных комбинаций атрибутов
Рендеринг может обрабатывать любую комбинацию пяти атрибутов, показанных выше.Ниже приведены несколько примеров рендеринга с различными комбинациями атрибутов.
| Дисплей | Описание |
|---|---|
| Уникальные, гармоничные, уникальные концы | |
| Множественный, согласованный, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, согласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, согласованный, один неуверенный конец, один виртуальный конец | |
| Уникальные, диссонирующие, уникальные концы | |
| Множественные, несогласованные, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, несогласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, противоречивый, один конец, не зависящий от уверенности, один виртуальный конец | |
| Уникальный, согласование не установлено, уникальные концы | |
| Множественные, согласование не установлено, один уникальный конец, один множественный конец | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец достоверности не установлена | |
| Уникальный, согласование не установлено, один конец с неопределенной достоверностью, один виртуальный конец | |
| Множественные, несогласованные, один уникальный конец, один множественный конец, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец с неопределенностью уверенности, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальные, согласованные, уникальные концы, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Клон без конца, без нити, уникальный, соответствие не установлено | |
| Клон без конца, плюс нить, уникальный, согласованный | |
| Клон без конца, без нити, множественный, соответствие не установлено | |
| Клон без конца, плюс нить, несогласованный, уникальный, не установлен |
4.Характеристики SNP
4.1 Цветовой код для дорожек SNP
В этой таблице описаны цвета по умолчанию для функций SNP. Эта окраска применяется к дорожкам dbSNP из базы данных NCBI SNP, а также к внешним / потоковым или загруженным файлам VCF.
Подробнее о рендеринге для треков ClinVar или GWAS см. Ниже.
| Тип модификации | Цвет |
|---|---|
| Однонуклеотидная вариация (SNV) | Красный |
| Удаление / вставка (DELINS) | Сине-фиолетовый |
| Удаление (DEL) | Синий |
| Вставка (INS) | желтый |
| Множественные вариации нуклеотидов (MNV) | Светло-серый |
4.1.1 Визуальные примеры
4.1.2 Вставка или удаление SNP
Дополнительное обозначение различает вставки и удаления.
Вставки отмечены двумя треугольниками в виде песочных часов:
Исключения отмечены треугольником, направленным вниз:
4.2 Визуализация треков ClinVar
дорожек данных VCF из проекта NCBI ClinVar имеют четкую окраску и визуализацию.
| Интерпретация ClinVar | Цвет |
|---|---|
| Патогенное | Темно-пурпурный |
| Вероятно патогенный | розовый |
| Доброкачественные | Темно-зеленый |
| Скорее всего доброкачественная | зеленый |
| Ответ на лекарства | Синий |
| Не предусмотрено; Неопределенное значение; Противоречивые интерпретации; Все остальные статусы ClinVar | Темно-серый |
Метки треков указывают на принадлежность вариации ClinVar и аллель ClinVar.
Дополнительную информацию, включая статус обзора ClinVar и прогнозируемые молекулярные последствия, можно найти во всплывающих подсказках к функциям.
4.3 Визуализация результатов общегеномной ассоциации
В этом разделе описывается отрисовка для анализа полногеномных ассоциаций (GWAS) из NCBI dbGaP.
Цвет представляет наивысшее значение p в этой ячейке.
| Диапазон значений p | Цвет |
|---|---|
| <2 | бирюзовый |
| 2–3 | Небесно-голубой |
| 3-4 | Синий |
| 4-5 | зеленый |
| 5-6 | желтый |
| 6-7 | Оранжевый |
| > 7 | Красный |
График разброса над ячейками отражает отдельные значения p.И цвет (который использует ту же цветовую схему), и расположение точки на оси Y соответствуют значению p.
5. Варианты конструкции
В этом разделе описывается отрисовка для структурных вариантов с идентификаторами NCBI dbVar.
5.1 Общий рендеринг
Существует четыре общих сценария для большинства вариантов (либо SV, либо SSV), как показано в таблице ниже. Однако также возможны смешанные случаи с определенной точкой останова на одном конце и неопределенным диапазоном точек останова на другом конце.Здесь мы используем (CNV SV) в качестве примера:
| Тип точки останова | Рендеринг | Визуальный пример |
|---|---|---|
| С разрешением точки останова | Полностью насыщенный цвет | |
| С определенным диапазоном точки останова | Прозрачный цвет для диапазонов контрольных точек | |
| С неопределенной точкой останова, но известной внешней границей | Треугольники, направленные друг к другу | |
| С неопределенной точкой останова, но известной внутренней границей | Треугольники, направленные друг от друга |
5.2 варианта типов вызовов (SSV) и типы регионов (SV)
| Тип | Комментарий | Визуальный пример |
|---|---|---|
| Изменение количества копий | Цвет: фиолетовый Четыре общих случая плюс CNV с длиной делеции (CNV) | |
| Увеличение числа копий или дублирование | Цвет: синий (усиление SSV) | |
| Утеря или удаление номера копии | Цвет: красный Последний вариант потери с длиной делеции (SSV потери) | |
| Вставка мобильного элемента или Вставка новой последовательности | Цвет: синий (вставка SV или SSV) | |
| Тандемное дублирование | Цвет: темно-коричневый (Eversion SV или SSV) | |
| инверсия | Цвет: светло-фиолетовый (инверсия SV или SSV) | |
| Перемещение | Цвет: светлый индиго с рисунком (Translocation SV или SSV) | |
| Комплекс | Цвет: светло-лазурный (Комплекс SSV) | |
| Вставка / удаление или отступление | Цвет: красный | |
| Неизвестно | Цвет: серый (Неизвестный SV или SSV) |
Примечание:
- Тип области SV «Вариант количества копий» может иметь дочерние элементы только для типов SSV «Увеличение количества копий» и / или «Потеря количества копий» — в любой комбинации.Если все дочерние SSV данного SV представляют собой увеличение количества копий или потерю количества копий, тогда SV будет окрашен так же, как окрашены дочерние элементы: синий для усиления и красный для потери, соответственно. Если дети представляют собой смесь этих двух типов, то SV будет окрашен в фиолетовый цвет.
СВ типа «Комплекс» может иметь:
детей всех ССВ типа «Комплекс», или
- дочерних элементов двух или более типов SSV в любой комбинации (за исключением «увеличения числа копий» и «потери числа копий», которые описаны выше)
5.3. Группа стилей визуализации для связанных структурных вариантов
5.3.1 Отрисовка по умолчанию с отображением родительского и дочернего элементов
5.3.2 Визуализация с опорными вариантами в упакованном виде
Если в поддерживающих вариантах имеется несколько типов, будет использоваться несколько цветов, чтобы отразить соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все поддерживаемые варианты.
5.3.3 Наложение всех поддерживающих вариантов на родительский вариант
Опорные варианты накладываются поверх родительского варианта, а самые короткие варианты — сверху. Цвета отражают соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все варианты.
5,4. Интерактивная визуализация дочерних вызовов в альтернативной области
Установить параметры рендеринга варианта Вкладка диалогового окна Настройка как «Показать родительский элемент, развернуть дочерние элементы по щелчку».
Sequence Viewer покажет знак плюса с количеством дочерних дорожек для каждой родительской дорожки.
Щелкните значок «плюс», и программа просмотра последовательности покажет все дочерние дорожки и отобразит знак минус вместо плюса на родительской дорожке.
Щелкните значок «минус», чтобы снова свернуть дочерние дорожки.
6. Сегментарное дублирование
| Атрибут идентификатора | Цвет | Пример |
|---|---|---|
| > 99.0 | Оранжевый | |
| > 98,0 | желтый | |
| > 90,0 | Серый | |
| <= 90,0 | Черный |
7. Выравнивания
Дорожки выравнивания содержат дискретные последовательности, попарно выровненные по отношению к сборке верхнего уровня. Эти треки получены из выравниваний последовательностей, представленных в BAM или аналогичных форматах.Выравнивание сборки-сборки и результаты NCBI BLAST также визуализируются в виде дорожек выравнивания в графическом представлении последовательности.
7.1 Дисплей выравнивания
Когда для параметра «Отображение выравнивания » установлено значение «Адаптивное» (по умолчанию), данные выравнивания представляются по-разному при разных уровнях масштабирования (рисунок 7.1.1). Отображение выравнивания также можно установить в режим «Упаковано» (всегда показывает покрытие или представление в виде скоплений), «Лестничная диаграмма» (одно выравнивание на строку) и «Показать все» (всегда показывает отдельные выравнивания).Эти параметры находятся в меню настроек отображения трека (Рисунок 7.3.1 и Таблица 7.3).
Рисунок 7.1.1 . Просмотр совмещений с различными уровнями масштабирования, когда Отображение выравнивания установлено на «Адаптивный». При более низких уровнях масштабирования выравнивания отображаются в виде графиков покрытия или наложения в масштабе 2 журнала. При более высоких уровнях масштабирования становятся видны отдельные выравнивания. Обратите внимание, что пробелы и несоответствия окрашены в красный цвет.
При высоких уровнях масштабирования становятся очевидными детали выравнивания.По умолчанию совпадающие базы и интроны окрашены в серый цвет, несоответствия и пробелы — в красный, а вставки представлены синими полосами (при более высоких уровнях масштабирования) или синими песочными часами с вертикальными синими полосами, пропорциональными количеству вставленных баз (рис. 7.1.2). Знаки «меньше» или «больше» (<или>) указывают ориентацию выравнивания относительно сборки.
Рисунок 7.1.2 . Легенда об особенностях центровки.
7.2 Подсказки по выравниванию
При наведении курсора на дискретное выравнивание появляется всплывающая подсказка, в которой приводится дополнительная информация о выравнивании (рисунок 7.2.1). Всплывающие подсказки сообщают о покрытии, идентичности и количестве несовпадений, интронов, невыровненных областей и пробелов в выравнивании. Покрытие рассчитывается как количество выровненных оснований, деленное на общую длину последовательности признаков, в то время как идентичность рассчитывается как количество совпадающих оснований, деленное на выровненную длину (исключая пробелы или интроны, без штрафа за пробелы).
Если возможно / применимо, также предоставляется строка CIGAR. Также есть ссылка на последовательность FASTA исходного выровненного объекта.Кроме того, невыровненные нуклеотиды 5 ‘и 3’ указаны в нижней части всплывающей подсказки, и их можно просмотреть, нажав кнопку «Просмотр».
Когда всплывающая подсказка создается рядом с положением вставки (вставок) в выравнивании, информация о вставке отображается в разделе «Невыровненные области» всплывающей подсказки. Все координаты сообщаются относительно выровненного объекта. В одной всплывающей подсказке сообщается до трех вставок.
Всплывающую подсказку можно «закрепить» с помощью канцелярской кнопки в верхнем левом углу.Вся информация, представленная во всплывающей подсказке, может быть выделена и скопирована в буфер обмена. Щелчок по лупе вверху сбрасывает графическое представление выбранного объекта.
Рисунок 7.2.1 . Всплывающая подсказка для функции выравнивания, сообщающей о покрытии, идентичности и количестве несоответствий, пропусков и невыровненных областей в выравнивании. Последовательность и координаты невыровненных регионов доступны в нижней части всплывающей подсказки. Информация для вставки (вставок) предоставляется, если всплывающая подсказка создается в позиции вставки.
7.3 Параметры отображения выравнивания
Доступ к настройкам отображения трека можно получить, щелкнув название трека или выбрав опцию Configure Tracks под кнопкой Tracks на панели инструментов. Меню настроек отображения треков (рисунок 7.3.1) для треков совмещения предоставляет множество различных вариантов отображения и анализа (таблица 7.3). Эти параметры могут включать в себя создание представления наложения, проецирование элементов (рисунок 7.3.5) и связывание пар сопряжений (рисунок 7.3.6). Когда выбрана опция Показать pileup , над трассой появляется представление pileup (рисунок 7.3.2). Раскрывающееся меню Pileup Display включает параметры для наложения, включая отображение графика совпадений / несоответствий (вид по умолчанию, рисунок 7.3.2), таблицы совпадений / несоответствий (рисунок 7.3.3) или таблицы ATGC (рисунок 7.3.2). 4). Выпадающее меню Unaligned Tails включает параметры для отображения / скрытия невыровненных длин хвоста и невыровненных последовательностей. Для треков, для которых доступны метаданные, пользователи также могут скрыть выравнивания (дубликаты, плохие чтения) (рисунок 7.3.7) или отсортируйте совпадения по цепям или гаплотипам.
Рисунок 7.3.1 . Настройки отображения треков для трасс выравнивания.
Дополнительные параметры отображения доступны для Выравнивание сборки и сборки дорожек, предоставленных NCBI. Эти дорожки состоят из выравниваний первого и второго прохода, которые по умолчанию будут отображаться на виде. Выравнивания с первым проходом представляют собой последовательности, которые имеют взаимное наилучшее выравнивание на обеих сборках (оценка взаимности 3).Выравнивания во втором проходе обеспечивают лучшее выравнивание на одной из сборок в другом месте (оценка взаимности 1 или 2). Раскрывающееся меню Сортировка выравниваний для дорожек выравнивания сборки-сборки содержит дополнительную опцию для сортировки вида дорожки по оценке взаимности (первый проход по сравнению со вторым проходом). Выравнивания второго прохода также можно скрыть из поля зрения, сняв флажок Показать выравнивания второго прохода (по умолчанию установлено). Оценка взаимности для дорожек выравнивания сборки-сборки также указывается во всплывающих подсказках отдельных выравниваний.
| Категория отображения | Параметры раскрывающегося меню |
|---|---|
| Отображение выравнивания | Adaptive Packed Лестница (одно выравнивание на ряд) Показать все |
| Pileup Display | График совпадения / несоответствия (количество) График совпадения / несоответствия (процент) График ATGC (количество) График ATGC (процент) Таблица совпадений / несоответствий (количество) Таблица совпадений / несоответствий (процент) Таблица ATGC (количество) Таблица ATGC (в процентах) |
| Метод оценки * | Оценка качества колонки ДНК Разница на основе частоты Показать различия Цвета нуклеиновых кислот Показатель качества Отключено |
| Невыровненные хвосты | Скрыть Показать длину хвоста Показать последовательность |
| Скрыть выравнивания * | Нет Дубликаты Плохие чтения Дубликаты / Плохие чтения |
| Сортировать выравнивания по * | Без сортировки Нить выравнивания Гаплотип Оценка взаимности |
Таблица 7.3 . Параметры отображения выравнивания находятся в раскрывающихся меню в меню настроек отображения трека. * Эти параметры могут быть неполными и могут различаться в зависимости от характера данных основного трека.
Рисунок 7.3.2 . Когда для параметра Pileup Display установлено значение «График совпадения / несоответствия» (по умолчанию), над выравниванием отображается график наложения. Красные отметки на графике обозначают несовпадения, а черные отметки обозначают пробелы. Отображение центровки на рисунке усечено.
Рисунок 7.3.3 . Когда для параметра Pileup Display установлено значение «Таблица соответствия / несоответствия (количество)», над выравниванием отображается таблица наложения. Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается счетчик интрона, пробела, несоответствия, совпадения и общего количества. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.4 . Когда Pileup Display установлен на «Таблица ATGC (счетчик)», таблица наложения отображается над выравниванием.Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается общее количество, A, T, G, C, пробел и интрон. Каждый нуклеотид представлен разным цветом; интенсивность цвета коррелирует с долей совпадений с этим нуклеотидом в этой позиции. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.5 . Функция выравнивания RefSeqGene (NG_029920.1) с выбранной опцией Project features показывает аннотированный транскрипт RefSeq (NM_), белок (NP_) и другие функции.
Рисунок 7.3.6 . Когда выбрана опция Link Mate Pairs , пары сопряжения последовательности, если это указано метаданными в дорожке, связаны красной пунктирной линией на дисплее выравнивания. Когда выбран параметр Показать метки , отображаются метки для пар сопряжений.
Рисунок 7.3.7 . Дубликаты ПЦР, если они указаны метаданными на дорожке, окрашиваются в фиолетовый цвет. Дубликаты ПЦР также могут быть указаны во всплывающей подсказке.Дублирующиеся трассы можно скрыть от просмотра, выбрав «Дубликаты» в раскрывающемся меню Скрыть трассы .
8. Дорожка последовательности
Дорожка последовательности представлена сплошной полосой при высоких уровнях масштабирования. Нажатие кнопки увеличения для перехода к последовательности (обозначено стрелкой ниже) изменяет уровень масштабирования для отображения последовательности.
Для нуклеотидных последовательностей показаны как первичная, так и комплементарная цепи. Метки 5 ’(обведены синим кружком) на каждом конце дорожки указывают ориентацию прядей.
По умолчанию дорожка последовательности расположена чуть ниже линейки, а метка дорожки скрыта. Положение дорожки можно изменить с помощью перетаскивания.
Настройка отображения дорожки для дорожки последовательности доступна на панели конфигурации дорожки и предоставляет возможность отображать метку для этой дорожки и цветовые промежутки по типу промежутка.
Если установлен флажок Цветовые промежутки по типу , промежутки будут окрашены в соответствии с аннотацией к соответствующему доступу INSDC для последовательности (т.е. аннотацию к записи плоского файла GenBank). Зазоры известной длины будут окрашены в фиолетовый цвет, зазоры неизвестной длины — в красный цвет, а загрязнения — в желтый.
8.1 Цветовой код сегмента
Для собранных последовательностей (например, геномных сборок) разные типы сегментов последовательности будут окрашены по-разному в зависимости от природы лежащего в основе компонента.
| Тип сегмента | Готовый HTGS | Осадка HTGS | WGS | Другое | Разрыв |
|---|---|---|---|---|---|
| Цвет | Синий | Оранжевый | зеленый | Серый | Черный |
Сегмент промежутка может быть другого цвета, кроме черного, если установлен флажок Цветовые промежутки по типу (см. Выше).
8.2 Ограничение Карта сайта
Карты распознавания рестрикционного фермента, например MAP_000012.1, можно рассматривать как треки последовательности с сайтами рестрикции, обозначенными зелеными треугольниками под полосой последовательности.
| Посмотреть | Визуальный пример |
|---|---|
| Увеличенное изображение | |
| Увеличенное изображение с всплывающей подсказкой (при наведении курсора мыши на сайт) |
9.Карта сегмента
Дорожки данных каркасов (контигов) и траекторий (компонентов) можно найти в группе вертикальных вкладок Sequence на панели конфигурации дорожек.
9.1 Подмости
9.2 Путь мозаики (компоненты)
Раскраска элементов траектории мозаики соответствует цветовой схеме из Раздела 8.1. Перекрывающиеся области, не входящие в контур, окрашены в желтый цвет.
10. Шесть кадровых трансляций
11.Размещение этикетки
Существует четыре глобальных варианта размещения меток: по умолчанию, боковая метка, верхняя метка и без метки. «По умолчанию» может означать разные настройки для разных объектов. Например, метка по умолчанию для трасс — это метка сверху, а настройка по умолчанию для элементов — это метка сбоку.
11.1 Боковая этикетка по сравнению с прядью
В режиме боковой надписи этикетка всегда размещается на стороне объекта размером 5 футов.
11.2 Примеры
12. Отрисовка гистограммы или графика
Гистограмма или графическое представление используется для визуализации данных, которые распределяются в виде количества данных на позицию.Эти данные часто представлены в формате Wig, bigWig или bedGraph. Типы данных, отображаемые в виде графиков, включают данные последовательности РНК, эпигеномные данные и данные о сохранении. Настройки отображения трека позволяют пользователям выбирать между стилями отображения («Гистограмма», «Тепловая карта», «Линейный график») и масштабом («Линейный», «Лог-база 10», «Лог-база e», «Лог-база 2») .
По умолчанию диапазон значений гистограммы или линейного графика соответствует локальным видимым минимальным и максимальным значениям на дисплее. Масштабирование, панорамирование или иное изменение отображаемой области приведет к соответствующей настройке минимального и максимального значений.
Пользователь может выбрать опцию Set Value Range для ручного управления диапазоном значений на дисплее. Этот параметр доступен, если для стиля отображения задано значение «Гистограмма» или «Линейный график», а для масштаба установлено значение «Линейный». Если поставщик данных установил минимальное и максимальное значения для трека, поля Min value и Max value будут предварительно заполнены этими значениями. Пользователь может применять предварительно заполненные диапазоны значений или настраивать диапазон значений вручную.Если трек является частью составной группы треков из концентратора треков, доступна опция массового применения настроек ко всем отображаемым трекам в составной группе.
После применения пользовательских минимальных и максимальных значений любые данные графика, выходящие за пределы установленного диапазона значений, будут обрезаны на дисплее и окрашены в красный цвет, чтобы предупредить пользователя. Когда параметр Set Value Range не выбран, диапазон значений вернется к использованию локального минимума и максимума на дисплее.
12.1 трек-концентраторы: multiWig Tracks
Некоторые концентраторы треков включают файлы multiWig, которые состоят из нескольких субтреков. Файлы MultiWig отображаются на графическом дисплее в виде объединенных графиков с каждым субтреком разным цветом (рисунок 12.1.1). Настройки отображения дорожек для дорожек multiWig предоставляют параметры для настройки масштаба и отображения субтреков в сложенном или объединенном формате.
При нажатии на легенду субтрека открывается диалоговое окно настроек отображения субтрека (рисунок 12.1.2). Пользователи могут настроить цвет, непрозрачность и стиль отображения отдельных фрагментов, которые перечислены в раскрывающемся меню. Также есть возможность скрыть отдельные субтреки.
Рисунок 12.1.1. Объединенные и составные представления файлов multiWig. Щелкните легенду, чтобы получить доступ к настройкам отображения субтрека.
Рисунок 12.1.2. Диалог настроек отображения субтрека. Выпадающее меню обеспечивает доступ к настройкам для всех субтреков в треке multiWig.
Легенда графического представления
Эта легенда относится к программе просмотра последовательности NCBI. NCBI Genome Workbench использует аналогичные графические представления.
- Визуализация общих функций
- Особенности модели гена
- Особенности размещения клонов
- Характеристики SNP
- Варианты конструкции
- Сегментарные дублирования
- Раскладки
- Дорожка последовательности
- Карта сегмента
- Шесть кадровых переводов
- Размещение этикетки
- Отрисовка гистограммы или графика
1.Отрисовка общих функций
В этом разделе описывается отрисовка, используемая для функций генов, моделей РНК и белков, регуляторных сайтов и большинства других типов функций. Эти функции часто происходят из файлов, отформатированных как BED, bigBED, GFF3, GTF, ASN.1 и аналогичные форматы.
Специализированный рендеринг для SNP, структурных вариантов и сегментных дубликатов описан в следующих разделах этой документации.
1.1 Код цвета элемента
Белые стрелки внутри объектов указывают направление объекта относительно последовательности верхнего уровня.Серые стрелки в интронах (например, в характеристиках РНК и кодирующей области) указывают направление сплайсинга.
1.2 Параметры отображения функций
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks .
Пользователи могут установить разрешение дисплея с помощью раскрывающегося меню Параметры рендеринга . По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
Параметр , связывающий параметры , обеспечивает различные виды отображения для дорожек, содержащих родительские и дочерние функции; т.е. «Показать все»; «Показать родителя, объединить потомков»; «Показывать детей, а не родителей»; «Показывать родителей, а не детей»; «Наложите детей на родителя». Изменение этих параметров может привести к отображению большего или меньшего количества функций на дорожке, в зависимости от базового источника данных на дорожке.
1.2.1 Отображение функции интрона
Дорожкиинтрона отображают сплайсинговые соединения экзон-экзон на основе анализа данных RNA-seq. Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Дорожки функцийIntron содержат дополнительные параметры отображения дорожек, в том числе возможность сортировать элементы стыка сращивания по количеству считываний сращивания или по направлению сращивания (прядям).Пользователи также могут упростить отображение этой дорожки, фильтруя данные по количеству совмещенных считываний.
1.3 Специальные стили визуализации
1.3.1 Псевдогеновые признаки
| Настройки дисплея | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Показать все | Полосы в зеленой полосе гена | |
| Без генной полосы | Зеленая полоса фона | |
| Показать на одной строке со структурой экзона, без генной полосы | Зеленая полоса фона |
1.3.2 Элементы с несовершенным соответствием сборке
| Пример | Визуальный эффект | Визуальные примеры |
|---|---|---|
| РНК или CDS с несоответствием последовательности относительно сборки | Затененный фон |
1.3.3 Элементы с частичным расположением аннотаций (например, из-за ошибок сборки или зазоров)
| Примеры | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Частичный 5 ‘конец | Черные стрелки («<<» или «>>») на конце 5 ‘ | |
| Частичный 3 ‘конец | Черные стрелки («<<" или ">>») на конце 3 ‘ | |
| Внутренние неточные концы | Черные стрелки («<<» и «>>») на внутренних соединениях | |
| Частичные 5- и 3-дюймовые концы | Черные стрелки («<<» и «>>») на обоих концах |
1.3.4 Элемент, помеченный как частичный
| Примеры случаев | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Элемент, отмеченный как частичный на концах 5 ’и 3’ (белые стрелки, «<<» и «>>»), и элемент с частичным концом 3 ’(черные стрелки« >> ») | Белые стрелки («<<» и «>>») на обоих концах или черные стрелки («>>») на одном конце. |
1.3.5 Ограничение возможностей сайта
Сайты узнавания рестрикционного фермента помечены треугольными метками, указывающими сайты разрезов.Треугольные метки отсутствуют при высоких уровнях масштабирования. Когда элемент выбирается щелчком мыши, расположение участков разреза обозначается красными линиями волос.
| Посмотреть | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Сайт узнавания рестрикционного фермента | Треугольники указывают места разрезов | |
| Место ограничения (функция выбрана) | Треугольники указывают место разреза; линиями волос отмечаются границы пространственных объектов и участки выреза | |
| Место ограничения (функция выбрана, уменьшено) | Без треугольников; линиями волос отмечаются границы пространственных объектов и участки выреза |
1.4 украшения
| Стиль декора | Визуальный эффект | Визуальные примеры |
|---|---|---|
| По умолчанию | Сплошные столбцы для интервалов признаков или экзонов и сплошные линии для интронов | |
| Стрелки | Стрелки на обоих концах, показывающие нить, и заштрихованные полосы для интронов | |
| Квадратный анкер | Квадрат для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Круглый анкер | Круг для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Необычный | Круг для начала только мРНК, квадрат для начала других признаков, кроме гена и CDS, стрелка для остановки функции, заштрихованные столбцы для интронов мРНК и кривые линии для интронов CDS |
2.Особенности модели гена
Характеристики генной модели состоят из нескольких возможных компонентов: генная панель, РНК / мРНК, CDS и характеристики экзона.
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks . Для треков аннотации генов NCBI или Ensembl могут быть доступны несколько вариантов. Параметр Hide non-coding скроет некодирующие транскрипты или некодирующие гены или псевдогены, чтобы отображались только варианты транскриптов, кодирующие белок. Hide model transcripts Функция скрывает модели стенограммы, предсказанные с помощью алгоритма Gnomon NCBI.
У вас также могут быть опции для отображения только вариантов, которые являются частью проекта CCDS или были обозначены как вариант RefSeq или MANE Select.
2.1 Визуализация генной модели
Настройки отображения трека включают несколько опций рендеринга, как описано ниже.
Когда все функции развернуты, столбцы генов окрашиваются в зеленый цвет, некодирующие РНК и элементы мРНК окрашиваются в фиолетовый цвет, а элементы CDS — в красный цвет.
При слиянии пар транскрипта и CDS кодирующие экзоны отображаются темно-зеленым цветом, а некодирующие области (т. Е. 5 ‘и 3’ UTR) отображаются более светлым оттенком зеленого, а некодирующие РНК остаются пурпурными (см. Пример «Объединить транскрипт и пары CDS, без генной полосы»).
Когда все транскрипты объединены и объединенные функции включают части экзонов, которые присутствуют в некоторых транскриптах, но отсутствуют в других транскриптах, интенсивность зеленой окраски пропорциональна количеству вариантов транскриптов, которые включают конкретную область экзона (см. «Объединить все транскрипты и CDS, без генной полосы », пример ниже).
Чтобы показать признаки, аннотированные на РНК или CDS, установите флажок Показать аннотированные признаки белка .
2.2 Специальная визуализация расхождений между аннотированными последовательностями RefSeq и геномными последовательностями
Визуализация несовпадений
В случае несинонимичных несовпадений, несоответствие выделяется комбинацией красного текста для кодона, отображения альтернативных оснований в мРНК и отображения альтернативных аминокислотных остатков в белковой характеристике.
В случае синонимичных несоответствий несоответствие выделяется комбинацией красного текста для кодона и отображения альтернативных оснований в мРНК.
Обработка вставок и удалений
Делеции отображаются в виде штрихов в соответствующих характеристиках мРНК и белков и отсутствуют какие-либо кодоны или аминокислоты.
Вставки отображаются в виде пары перевернутых вертикальных треугольников в мРНК и характеристиках белка, и показаны соответствующие кодоны и аминокислоты; кодоны окрашены в красный цвет.
2.3 Функциональная линейка
Когда все признаки развернуты в дорожке генной модели, при выборе объекта РНК или CDS, щелкнув по нему, появится линейка признаков, указывающая локальные координаты признака.
Когда выбрано несколько элементов, появляются линии волос, указывающие на различия в структуре разных элементов.
2.4 Подсказка по функциям
При наведении курсора на объект появляется всплывающая подсказка с дополнительной информацией об этом объекте.Всплывающая подсказка для функций генов, РНК и CDS сообщает о длине элемента, размахе сборки и координатах позиций, в которых всплывающая подсказка была активирована. Также включены ссылки на другие NCBI или внешние ресурсы, относящиеся к гену или функции, если таковые имеются. Также есть варианты для загрузки функции в форматах FASTA или GenBank, отправки ее в BLAST или открытия в ее собственном экземпляре графического средства просмотра последовательности. Более подробная информация о всплывающих подсказках для генов доступна здесь.
3. Особенности размещения клонов
3.1 Сообщаемые атрибуты
Графические изображения для всех элементов размещения клонов передают следующие атрибуты:
- Согласованность
- Уникальность
- Клонирование конца уверенности
- направленность и
- Подтверждающие доказательства
3.2 Визуальные примеры для переданного атрибута
3.3 Примеры визуализации для различных комбинаций атрибутов
Рендеринг может обрабатывать любую комбинацию пяти атрибутов, показанных выше.Ниже приведены несколько примеров рендеринга с различными комбинациями атрибутов.
| Дисплей | Описание |
|---|---|
| Уникальные, гармоничные, уникальные концы | |
| Множественный, согласованный, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, согласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, согласованный, один неуверенный конец, один виртуальный конец | |
| Уникальные, диссонирующие, уникальные концы | |
| Множественные, несогласованные, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, несогласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, противоречивый, один конец, не зависящий от уверенности, один виртуальный конец | |
| Уникальный, согласование не установлено, уникальные концы | |
| Множественные, согласование не установлено, один уникальный конец, один множественный конец | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец достоверности не установлена | |
| Уникальный, согласование не установлено, один конец с неопределенной достоверностью, один виртуальный конец | |
| Множественные, несогласованные, один уникальный конец, один множественный конец, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец с неопределенностью уверенности, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальные, согласованные, уникальные концы, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Клон без конца, без нити, уникальный, соответствие не установлено | |
| Клон без конца, плюс нить, уникальный, согласованный | |
| Клон без конца, без нити, множественный, соответствие не установлено | |
| Клон без конца, плюс нить, несогласованный, уникальный, не установлен |
4.Характеристики SNP
4.1 Цветовой код для дорожек SNP
В этой таблице описаны цвета по умолчанию для функций SNP. Эта окраска применяется к дорожкам dbSNP из базы данных NCBI SNP, а также к внешним / потоковым или загруженным файлам VCF.
Подробнее о рендеринге для треков ClinVar или GWAS см. Ниже.
| Тип модификации | Цвет |
|---|---|
| Однонуклеотидная вариация (SNV) | Красный |
| Удаление / вставка (DELINS) | Сине-фиолетовый |
| Удаление (DEL) | Синий |
| Вставка (INS) | желтый |
| Множественные вариации нуклеотидов (MNV) | Светло-серый |
4.1.1 Визуальные примеры
4.1.2 Вставка или удаление SNP
Дополнительное обозначение различает вставки и удаления.
Вставки отмечены двумя треугольниками в виде песочных часов:
Исключения отмечены треугольником, направленным вниз:
4.2 Визуализация треков ClinVar
дорожек данных VCF из проекта NCBI ClinVar имеют четкую окраску и визуализацию.
| Интерпретация ClinVar | Цвет |
|---|---|
| Патогенное | Темно-пурпурный |
| Вероятно патогенный | розовый |
| Доброкачественные | Темно-зеленый |
| Скорее всего доброкачественная | зеленый |
| Ответ на лекарства | Синий |
| Не предусмотрено; Неопределенное значение; Противоречивые интерпретации; Все остальные статусы ClinVar | Темно-серый |
Метки треков указывают на принадлежность вариации ClinVar и аллель ClinVar.
Дополнительную информацию, включая статус обзора ClinVar и прогнозируемые молекулярные последствия, можно найти во всплывающих подсказках к функциям.
4.3 Визуализация результатов общегеномной ассоциации
В этом разделе описывается отрисовка для анализа полногеномных ассоциаций (GWAS) из NCBI dbGaP.
Цвет представляет наивысшее значение p в этой ячейке.
| Диапазон значений p | Цвет |
|---|---|
| <2 | бирюзовый |
| 2–3 | Небесно-голубой |
| 3-4 | Синий |
| 4-5 | зеленый |
| 5-6 | желтый |
| 6-7 | Оранжевый |
| > 7 | Красный |
График разброса над ячейками отражает отдельные значения p.И цвет (который использует ту же цветовую схему), и расположение точки на оси Y соответствуют значению p.
5. Варианты конструкции
В этом разделе описывается отрисовка для структурных вариантов с идентификаторами NCBI dbVar.
5.1 Общий рендеринг
Существует четыре общих сценария для большинства вариантов (либо SV, либо SSV), как показано в таблице ниже. Однако также возможны смешанные случаи с определенной точкой останова на одном конце и неопределенным диапазоном точек останова на другом конце.Здесь мы используем (CNV SV) в качестве примера:
| Тип точки останова | Рендеринг | Визуальный пример |
|---|---|---|
| С разрешением точки останова | Полностью насыщенный цвет | |
| С определенным диапазоном точки останова | Прозрачный цвет для диапазонов контрольных точек | |
| С неопределенной точкой останова, но известной внешней границей | Треугольники, направленные друг к другу | |
| С неопределенной точкой останова, но известной внутренней границей | Треугольники, направленные друг от друга |
5.2 варианта типов вызовов (SSV) и типы регионов (SV)
| Тип | Комментарий | Визуальный пример |
|---|---|---|
| Изменение количества копий | Цвет: фиолетовый Четыре общих случая плюс CNV с длиной делеции (CNV) | |
| Увеличение числа копий или дублирование | Цвет: синий (усиление SSV) | |
| Утеря или удаление номера копии | Цвет: красный Последний вариант потери с длиной делеции (SSV потери) | |
| Вставка мобильного элемента или Вставка новой последовательности | Цвет: синий (вставка SV или SSV) | |
| Тандемное дублирование | Цвет: темно-коричневый (Eversion SV или SSV) | |
| инверсия | Цвет: светло-фиолетовый (инверсия SV или SSV) | |
| Перемещение | Цвет: светлый индиго с рисунком (Translocation SV или SSV) | |
| Комплекс | Цвет: светло-лазурный (Комплекс SSV) | |
| Вставка / удаление или отступление | Цвет: красный | |
| Неизвестно | Цвет: серый (Неизвестный SV или SSV) |
Примечание:
- Тип области SV «Вариант количества копий» может иметь дочерние элементы только для типов SSV «Увеличение количества копий» и / или «Потеря количества копий» — в любой комбинации.Если все дочерние SSV данного SV представляют собой увеличение количества копий или потерю количества копий, тогда SV будет окрашен так же, как окрашены дочерние элементы: синий для усиления и красный для потери, соответственно. Если дети представляют собой смесь этих двух типов, то SV будет окрашен в фиолетовый цвет.
СВ типа «Комплекс» может иметь:
детей всех ССВ типа «Комплекс», или
- дочерних элементов двух или более типов SSV в любой комбинации (за исключением «увеличения числа копий» и «потери числа копий», которые описаны выше)
5.3. Группа стилей визуализации для связанных структурных вариантов
5.3.1 Отрисовка по умолчанию с отображением родительского и дочернего элементов
5.3.2 Визуализация с опорными вариантами в упакованном виде
Если в поддерживающих вариантах имеется несколько типов, будет использоваться несколько цветов, чтобы отразить соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все поддерживаемые варианты.
5.3.3 Наложение всех поддерживающих вариантов на родительский вариант
Опорные варианты накладываются поверх родительского варианта, а самые короткие варианты — сверху. Цвета отражают соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все варианты.
5,4. Интерактивная визуализация дочерних вызовов в альтернативной области
Установить параметры рендеринга варианта Вкладка диалогового окна Настройка как «Показать родительский элемент, развернуть дочерние элементы по щелчку».
Sequence Viewer покажет знак плюса с количеством дочерних дорожек для каждой родительской дорожки.
Щелкните значок «плюс», и программа просмотра последовательности покажет все дочерние дорожки и отобразит знак минус вместо плюса на родительской дорожке.
Щелкните значок «минус», чтобы снова свернуть дочерние дорожки.
6. Сегментарное дублирование
| Атрибут идентификатора | Цвет | Пример |
|---|---|---|
| > 99.0 | Оранжевый | |
| > 98,0 | желтый | |
| > 90,0 | Серый | |
| <= 90,0 | Черный |
7. Выравнивания
Дорожки выравнивания содержат дискретные последовательности, попарно выровненные по отношению к сборке верхнего уровня. Эти треки получены из выравниваний последовательностей, представленных в BAM или аналогичных форматах.Выравнивание сборки-сборки и результаты NCBI BLAST также визуализируются в виде дорожек выравнивания в графическом представлении последовательности.
7.1 Дисплей выравнивания
Когда для параметра «Отображение выравнивания » установлено значение «Адаптивное» (по умолчанию), данные выравнивания представляются по-разному при разных уровнях масштабирования (рисунок 7.1.1). Отображение выравнивания также можно установить в режим «Упаковано» (всегда показывает покрытие или представление в виде скоплений), «Лестничная диаграмма» (одно выравнивание на строку) и «Показать все» (всегда показывает отдельные выравнивания).Эти параметры находятся в меню настроек отображения трека (Рисунок 7.3.1 и Таблица 7.3).
Рисунок 7.1.1 . Просмотр совмещений с различными уровнями масштабирования, когда Отображение выравнивания установлено на «Адаптивный». При более низких уровнях масштабирования выравнивания отображаются в виде графиков покрытия или наложения в масштабе 2 журнала. При более высоких уровнях масштабирования становятся видны отдельные выравнивания. Обратите внимание, что пробелы и несоответствия окрашены в красный цвет.
При высоких уровнях масштабирования становятся очевидными детали выравнивания.По умолчанию совпадающие базы и интроны окрашены в серый цвет, несоответствия и пробелы — в красный, а вставки представлены синими полосами (при более высоких уровнях масштабирования) или синими песочными часами с вертикальными синими полосами, пропорциональными количеству вставленных баз (рис. 7.1.2). Знаки «меньше» или «больше» (<или>) указывают ориентацию выравнивания относительно сборки.
Рисунок 7.1.2 . Легенда об особенностях центровки.
7.2 Подсказки по выравниванию
При наведении курсора на дискретное выравнивание появляется всплывающая подсказка, в которой приводится дополнительная информация о выравнивании (рисунок 7.2.1). Всплывающие подсказки сообщают о покрытии, идентичности и количестве несовпадений, интронов, невыровненных областей и пробелов в выравнивании. Покрытие рассчитывается как количество выровненных оснований, деленное на общую длину последовательности признаков, в то время как идентичность рассчитывается как количество совпадающих оснований, деленное на выровненную длину (исключая пробелы или интроны, без штрафа за пробелы).
Если возможно / применимо, также предоставляется строка CIGAR. Также есть ссылка на последовательность FASTA исходного выровненного объекта.Кроме того, невыровненные нуклеотиды 5 ‘и 3’ указаны в нижней части всплывающей подсказки, и их можно просмотреть, нажав кнопку «Просмотр».
Когда всплывающая подсказка создается рядом с положением вставки (вставок) в выравнивании, информация о вставке отображается в разделе «Невыровненные области» всплывающей подсказки. Все координаты сообщаются относительно выровненного объекта. В одной всплывающей подсказке сообщается до трех вставок.
Всплывающую подсказку можно «закрепить» с помощью канцелярской кнопки в верхнем левом углу.Вся информация, представленная во всплывающей подсказке, может быть выделена и скопирована в буфер обмена. Щелчок по лупе вверху сбрасывает графическое представление выбранного объекта.
Рисунок 7.2.1 . Всплывающая подсказка для функции выравнивания, сообщающей о покрытии, идентичности и количестве несоответствий, пропусков и невыровненных областей в выравнивании. Последовательность и координаты невыровненных регионов доступны в нижней части всплывающей подсказки. Информация для вставки (вставок) предоставляется, если всплывающая подсказка создается в позиции вставки.
7.3 Параметры отображения выравнивания
Доступ к настройкам отображения трека можно получить, щелкнув название трека или выбрав опцию Configure Tracks под кнопкой Tracks на панели инструментов. Меню настроек отображения треков (рисунок 7.3.1) для треков совмещения предоставляет множество различных вариантов отображения и анализа (таблица 7.3). Эти параметры могут включать в себя создание представления наложения, проецирование элементов (рисунок 7.3.5) и связывание пар сопряжений (рисунок 7.3.6). Когда выбрана опция Показать pileup , над трассой появляется представление pileup (рисунок 7.3.2). Раскрывающееся меню Pileup Display включает параметры для наложения, включая отображение графика совпадений / несоответствий (вид по умолчанию, рисунок 7.3.2), таблицы совпадений / несоответствий (рисунок 7.3.3) или таблицы ATGC (рисунок 7.3.2). 4). Выпадающее меню Unaligned Tails включает параметры для отображения / скрытия невыровненных длин хвоста и невыровненных последовательностей. Для треков, для которых доступны метаданные, пользователи также могут скрыть выравнивания (дубликаты, плохие чтения) (рисунок 7.3.7) или отсортируйте совпадения по цепям или гаплотипам.
Рисунок 7.3.1 . Настройки отображения треков для трасс выравнивания.
Дополнительные параметры отображения доступны для Выравнивание сборки и сборки дорожек, предоставленных NCBI. Эти дорожки состоят из выравниваний первого и второго прохода, которые по умолчанию будут отображаться на виде. Выравнивания с первым проходом представляют собой последовательности, которые имеют взаимное наилучшее выравнивание на обеих сборках (оценка взаимности 3).Выравнивания во втором проходе обеспечивают лучшее выравнивание на одной из сборок в другом месте (оценка взаимности 1 или 2). Раскрывающееся меню Сортировка выравниваний для дорожек выравнивания сборки-сборки содержит дополнительную опцию для сортировки вида дорожки по оценке взаимности (первый проход по сравнению со вторым проходом). Выравнивания второго прохода также можно скрыть из поля зрения, сняв флажок Показать выравнивания второго прохода (по умолчанию установлено). Оценка взаимности для дорожек выравнивания сборки-сборки также указывается во всплывающих подсказках отдельных выравниваний.
| Категория отображения | Параметры раскрывающегося меню |
|---|---|
| Отображение выравнивания | Adaptive Packed Лестница (одно выравнивание на ряд) Показать все |
| Pileup Display | График совпадения / несоответствия (количество) График совпадения / несоответствия (процент) График ATGC (количество) График ATGC (процент) Таблица совпадений / несоответствий (количество) Таблица совпадений / несоответствий (процент) Таблица ATGC (количество) Таблица ATGC (в процентах) |
| Метод оценки * | Оценка качества колонки ДНК Разница на основе частоты Показать различия Цвета нуклеиновых кислот Показатель качества Отключено |
| Невыровненные хвосты | Скрыть Показать длину хвоста Показать последовательность |
| Скрыть выравнивания * | Нет Дубликаты Плохие чтения Дубликаты / Плохие чтения |
| Сортировать выравнивания по * | Без сортировки Нить выравнивания Гаплотип Оценка взаимности |
Таблица 7.3 . Параметры отображения выравнивания находятся в раскрывающихся меню в меню настроек отображения трека. * Эти параметры могут быть неполными и могут различаться в зависимости от характера данных основного трека.
Рисунок 7.3.2 . Когда для параметра Pileup Display установлено значение «График совпадения / несоответствия» (по умолчанию), над выравниванием отображается график наложения. Красные отметки на графике обозначают несовпадения, а черные отметки обозначают пробелы. Отображение центровки на рисунке усечено.
Рисунок 7.3.3 . Когда для параметра Pileup Display установлено значение «Таблица соответствия / несоответствия (количество)», над выравниванием отображается таблица наложения. Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается счетчик интрона, пробела, несоответствия, совпадения и общего количества. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.4 . Когда Pileup Display установлен на «Таблица ATGC (счетчик)», таблица наложения отображается над выравниванием.Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается общее количество, A, T, G, C, пробел и интрон. Каждый нуклеотид представлен разным цветом; интенсивность цвета коррелирует с долей совпадений с этим нуклеотидом в этой позиции. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.5 . Функция выравнивания RefSeqGene (NG_029920.1) с выбранной опцией Project features показывает аннотированный транскрипт RefSeq (NM_), белок (NP_) и другие функции.
Рисунок 7.3.6 . Когда выбрана опция Link Mate Pairs , пары сопряжения последовательности, если это указано метаданными в дорожке, связаны красной пунктирной линией на дисплее выравнивания. Когда выбран параметр Показать метки , отображаются метки для пар сопряжений.
Рисунок 7.3.7 . Дубликаты ПЦР, если они указаны метаданными на дорожке, окрашиваются в фиолетовый цвет. Дубликаты ПЦР также могут быть указаны во всплывающей подсказке.Дублирующиеся трассы можно скрыть от просмотра, выбрав «Дубликаты» в раскрывающемся меню Скрыть трассы .
8. Дорожка последовательности
Дорожка последовательности представлена сплошной полосой при высоких уровнях масштабирования. Нажатие кнопки увеличения для перехода к последовательности (обозначено стрелкой ниже) изменяет уровень масштабирования для отображения последовательности.
Для нуклеотидных последовательностей показаны как первичная, так и комплементарная цепи. Метки 5 ’(обведены синим кружком) на каждом конце дорожки указывают ориентацию прядей.
По умолчанию дорожка последовательности расположена чуть ниже линейки, а метка дорожки скрыта. Положение дорожки можно изменить с помощью перетаскивания.
Настройка отображения дорожки для дорожки последовательности доступна на панели конфигурации дорожки и предоставляет возможность отображать метку для этой дорожки и цветовые промежутки по типу промежутка.
Если установлен флажок Цветовые промежутки по типу , промежутки будут окрашены в соответствии с аннотацией к соответствующему доступу INSDC для последовательности (т.е. аннотацию к записи плоского файла GenBank). Зазоры известной длины будут окрашены в фиолетовый цвет, зазоры неизвестной длины — в красный цвет, а загрязнения — в желтый.
8.1 Цветовой код сегмента
Для собранных последовательностей (например, геномных сборок) разные типы сегментов последовательности будут окрашены по-разному в зависимости от природы лежащего в основе компонента.
| Тип сегмента | Готовый HTGS | Осадка HTGS | WGS | Другое | Разрыв |
|---|---|---|---|---|---|
| Цвет | Синий | Оранжевый | зеленый | Серый | Черный |
Сегмент промежутка может быть другого цвета, кроме черного, если установлен флажок Цветовые промежутки по типу (см. Выше).
8.2 Ограничение Карта сайта
Карты распознавания рестрикционного фермента, например MAP_000012.1, можно рассматривать как треки последовательности с сайтами рестрикции, обозначенными зелеными треугольниками под полосой последовательности.
| Посмотреть | Визуальный пример |
|---|---|
| Увеличенное изображение | |
| Увеличенное изображение с всплывающей подсказкой (при наведении курсора мыши на сайт) |
9.Карта сегмента
Дорожки данных каркасов (контигов) и траекторий (компонентов) можно найти в группе вертикальных вкладок Sequence на панели конфигурации дорожек.
9.1 Подмости
9.2 Путь мозаики (компоненты)
Раскраска элементов траектории мозаики соответствует цветовой схеме из Раздела 8.1. Перекрывающиеся области, не входящие в контур, окрашены в желтый цвет.
10. Шесть кадровых трансляций
11.Размещение этикетки
Существует четыре глобальных варианта размещения меток: по умолчанию, боковая метка, верхняя метка и без метки. «По умолчанию» может означать разные настройки для разных объектов. Например, метка по умолчанию для трасс — это метка сверху, а настройка по умолчанию для элементов — это метка сбоку.
11.1 Боковая этикетка по сравнению с прядью
В режиме боковой надписи этикетка всегда размещается на стороне объекта размером 5 футов.
11.2 Примеры
12. Отрисовка гистограммы или графика
Гистограмма или графическое представление используется для визуализации данных, которые распределяются в виде количества данных на позицию.Эти данные часто представлены в формате Wig, bigWig или bedGraph. Типы данных, отображаемые в виде графиков, включают данные последовательности РНК, эпигеномные данные и данные о сохранении. Настройки отображения трека позволяют пользователям выбирать между стилями отображения («Гистограмма», «Тепловая карта», «Линейный график») и масштабом («Линейный», «Лог-база 10», «Лог-база e», «Лог-база 2») .
По умолчанию диапазон значений гистограммы или линейного графика соответствует локальным видимым минимальным и максимальным значениям на дисплее. Масштабирование, панорамирование или иное изменение отображаемой области приведет к соответствующей настройке минимального и максимального значений.
Пользователь может выбрать опцию Set Value Range для ручного управления диапазоном значений на дисплее. Этот параметр доступен, если для стиля отображения задано значение «Гистограмма» или «Линейный график», а для масштаба установлено значение «Линейный». Если поставщик данных установил минимальное и максимальное значения для трека, поля Min value и Max value будут предварительно заполнены этими значениями. Пользователь может применять предварительно заполненные диапазоны значений или настраивать диапазон значений вручную.Если трек является частью составной группы треков из концентратора треков, доступна опция массового применения настроек ко всем отображаемым трекам в составной группе.
После применения пользовательских минимальных и максимальных значений любые данные графика, выходящие за пределы установленного диапазона значений, будут обрезаны на дисплее и окрашены в красный цвет, чтобы предупредить пользователя. Когда параметр Set Value Range не выбран, диапазон значений вернется к использованию локального минимума и максимума на дисплее.
12.1 трек-концентраторы: multiWig Tracks
Некоторые концентраторы треков включают файлы multiWig, которые состоят из нескольких субтреков. Файлы MultiWig отображаются на графическом дисплее в виде объединенных графиков с каждым субтреком разным цветом (рисунок 12.1.1). Настройки отображения дорожек для дорожек multiWig предоставляют параметры для настройки масштаба и отображения субтреков в сложенном или объединенном формате.
При нажатии на легенду субтрека открывается диалоговое окно настроек отображения субтрека (рисунок 12.1.2). Пользователи могут настроить цвет, непрозрачность и стиль отображения отдельных фрагментов, которые перечислены в раскрывающемся меню. Также есть возможность скрыть отдельные субтреки.
Рисунок 12.1.1. Объединенные и составные представления файлов multiWig. Щелкните легенду, чтобы получить доступ к настройкам отображения субтрека.
Рисунок 12.1.2. Диалог настроек отображения субтрека. Выпадающее меню обеспечивает доступ к настройкам для всех субтреков в треке multiWig.
Легенда графического представления
Эта легенда относится к программе просмотра последовательности NCBI. NCBI Genome Workbench использует аналогичные графические представления.
- Визуализация общих функций
- Особенности модели гена
- Особенности размещения клонов
- Характеристики SNP
- Варианты конструкции
- Сегментарные дублирования
- Раскладки
- Дорожка последовательности
- Карта сегмента
- Шесть кадровых переводов
- Размещение этикетки
- Отрисовка гистограммы или графика
1.Отрисовка общих функций
В этом разделе описывается отрисовка, используемая для функций генов, моделей РНК и белков, регуляторных сайтов и большинства других типов функций. Эти функции часто происходят из файлов, отформатированных как BED, bigBED, GFF3, GTF, ASN.1 и аналогичные форматы.
Специализированный рендеринг для SNP, структурных вариантов и сегментных дубликатов описан в следующих разделах этой документации.
1.1 Код цвета элемента
Белые стрелки внутри объектов указывают направление объекта относительно последовательности верхнего уровня.Серые стрелки в интронах (например, в характеристиках РНК и кодирующей области) указывают направление сплайсинга.
1.2 Параметры отображения функций
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks .
Пользователи могут установить разрешение дисплея с помощью раскрывающегося меню Параметры рендеринга . По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
Параметр , связывающий параметры , обеспечивает различные виды отображения для дорожек, содержащих родительские и дочерние функции; т.е. «Показать все»; «Показать родителя, объединить потомков»; «Показывать детей, а не родителей»; «Показывать родителей, а не детей»; «Наложите детей на родителя». Изменение этих параметров может привести к отображению большего или меньшего количества функций на дорожке, в зависимости от базового источника данных на дорожке.
1.2.1 Отображение функции интрона
Дорожкиинтрона отображают сплайсинговые соединения экзон-экзон на основе анализа данных RNA-seq. Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Дорожки функцийIntron содержат дополнительные параметры отображения дорожек, в том числе возможность сортировать элементы стыка сращивания по количеству считываний сращивания или по направлению сращивания (прядям).Пользователи также могут упростить отображение этой дорожки, фильтруя данные по количеству совмещенных считываний.
1.3 Специальные стили визуализации
1.3.1 Псевдогеновые признаки
| Настройки дисплея | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Показать все | Полосы в зеленой полосе гена | |
| Без генной полосы | Зеленая полоса фона | |
| Показать на одной строке со структурой экзона, без генной полосы | Зеленая полоса фона |
1.3.2 Элементы с несовершенным соответствием сборке
| Пример | Визуальный эффект | Визуальные примеры |
|---|---|---|
| РНК или CDS с несоответствием последовательности относительно сборки | Затененный фон |
1.3.3 Элементы с частичным расположением аннотаций (например, из-за ошибок сборки или зазоров)
| Примеры | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Частичный 5 ‘конец | Черные стрелки («<<» или «>>») на конце 5 ‘ | |
| Частичный 3 ‘конец | Черные стрелки («<<" или ">>») на конце 3 ‘ | |
| Внутренние неточные концы | Черные стрелки («<<» и «>>») на внутренних соединениях | |
| Частичные 5- и 3-дюймовые концы | Черные стрелки («<<» и «>>») на обоих концах |
1.3.4 Элемент, помеченный как частичный
| Примеры случаев | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Элемент, отмеченный как частичный на концах 5 ’и 3’ (белые стрелки, «<<» и «>>»), и элемент с частичным концом 3 ’(черные стрелки« >> ») | Белые стрелки («<<» и «>>») на обоих концах или черные стрелки («>>») на одном конце. |
1.3.5 Ограничение возможностей сайта
Сайты узнавания рестрикционного фермента помечены треугольными метками, указывающими сайты разрезов.Треугольные метки отсутствуют при высоких уровнях масштабирования. Когда элемент выбирается щелчком мыши, расположение участков разреза обозначается красными линиями волос.
| Посмотреть | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Сайт узнавания рестрикционного фермента | Треугольники указывают места разрезов | |
| Место ограничения (функция выбрана) | Треугольники указывают место разреза; линиями волос отмечаются границы пространственных объектов и участки выреза | |
| Место ограничения (функция выбрана, уменьшено) | Без треугольников; линиями волос отмечаются границы пространственных объектов и участки выреза |
1.4 украшения
| Стиль декора | Визуальный эффект | Визуальные примеры |
|---|---|---|
| По умолчанию | Сплошные столбцы для интервалов признаков или экзонов и сплошные линии для интронов | |
| Стрелки | Стрелки на обоих концах, показывающие нить, и заштрихованные полосы для интронов | |
| Квадратный анкер | Квадрат для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Круглый анкер | Круг для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Необычный | Круг для начала только мРНК, квадрат для начала других признаков, кроме гена и CDS, стрелка для остановки функции, заштрихованные столбцы для интронов мРНК и кривые линии для интронов CDS |
2.Особенности модели гена
Характеристики генной модели состоят из нескольких возможных компонентов: генная панель, РНК / мРНК, CDS и характеристики экзона.
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks . Для треков аннотации генов NCBI или Ensembl могут быть доступны несколько вариантов. Параметр Hide non-coding скроет некодирующие транскрипты или некодирующие гены или псевдогены, чтобы отображались только варианты транскриптов, кодирующие белок. Hide model transcripts Функция скрывает модели стенограммы, предсказанные с помощью алгоритма Gnomon NCBI.
У вас также могут быть опции для отображения только вариантов, которые являются частью проекта CCDS или были обозначены как вариант RefSeq или MANE Select.
2.1 Визуализация генной модели
Настройки отображения трека включают несколько опций рендеринга, как описано ниже.
Когда все функции развернуты, столбцы генов окрашиваются в зеленый цвет, некодирующие РНК и элементы мРНК окрашиваются в фиолетовый цвет, а элементы CDS — в красный цвет.
При слиянии пар транскрипта и CDS кодирующие экзоны отображаются темно-зеленым цветом, а некодирующие области (т. Е. 5 ‘и 3’ UTR) отображаются более светлым оттенком зеленого, а некодирующие РНК остаются пурпурными (см. Пример «Объединить транскрипт и пары CDS, без генной полосы»).
Когда все транскрипты объединены и объединенные функции включают части экзонов, которые присутствуют в некоторых транскриптах, но отсутствуют в других транскриптах, интенсивность зеленой окраски пропорциональна количеству вариантов транскриптов, которые включают конкретную область экзона (см. «Объединить все транскрипты и CDS, без генной полосы », пример ниже).
Чтобы показать признаки, аннотированные на РНК или CDS, установите флажок Показать аннотированные признаки белка .
2.2 Специальная визуализация расхождений между аннотированными последовательностями RefSeq и геномными последовательностями
Визуализация несовпадений
В случае несинонимичных несовпадений, несоответствие выделяется комбинацией красного текста для кодона, отображения альтернативных оснований в мРНК и отображения альтернативных аминокислотных остатков в белковой характеристике.
В случае синонимичных несоответствий несоответствие выделяется комбинацией красного текста для кодона и отображения альтернативных оснований в мРНК.
Обработка вставок и удалений
Делеции отображаются в виде штрихов в соответствующих характеристиках мРНК и белков и отсутствуют какие-либо кодоны или аминокислоты.
Вставки отображаются в виде пары перевернутых вертикальных треугольников в мРНК и характеристиках белка, и показаны соответствующие кодоны и аминокислоты; кодоны окрашены в красный цвет.
2.3 Функциональная линейка
Когда все признаки развернуты в дорожке генной модели, при выборе объекта РНК или CDS, щелкнув по нему, появится линейка признаков, указывающая локальные координаты признака.
Когда выбрано несколько элементов, появляются линии волос, указывающие на различия в структуре разных элементов.
2.4 Подсказка по функциям
При наведении курсора на объект появляется всплывающая подсказка с дополнительной информацией об этом объекте.Всплывающая подсказка для функций генов, РНК и CDS сообщает о длине элемента, размахе сборки и координатах позиций, в которых всплывающая подсказка была активирована. Также включены ссылки на другие NCBI или внешние ресурсы, относящиеся к гену или функции, если таковые имеются. Также есть варианты для загрузки функции в форматах FASTA или GenBank, отправки ее в BLAST или открытия в ее собственном экземпляре графического средства просмотра последовательности. Более подробная информация о всплывающих подсказках для генов доступна здесь.
3. Особенности размещения клонов
3.1 Сообщаемые атрибуты
Графические изображения для всех элементов размещения клонов передают следующие атрибуты:
- Согласованность
- Уникальность
- Клонирование конца уверенности
- направленность и
- Подтверждающие доказательства
3.2 Визуальные примеры для переданного атрибута
3.3 Примеры визуализации для различных комбинаций атрибутов
Рендеринг может обрабатывать любую комбинацию пяти атрибутов, показанных выше.Ниже приведены несколько примеров рендеринга с различными комбинациями атрибутов.
| Дисплей | Описание |
|---|---|
| Уникальные, гармоничные, уникальные концы | |
| Множественный, согласованный, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, согласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, согласованный, один неуверенный конец, один виртуальный конец | |
| Уникальные, диссонирующие, уникальные концы | |
| Множественные, несогласованные, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, несогласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, противоречивый, один конец, не зависящий от уверенности, один виртуальный конец | |
| Уникальный, согласование не установлено, уникальные концы | |
| Множественные, согласование не установлено, один уникальный конец, один множественный конец | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец достоверности не установлена | |
| Уникальный, согласование не установлено, один конец с неопределенной достоверностью, один виртуальный конец | |
| Множественные, несогласованные, один уникальный конец, один множественный конец, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец с неопределенностью уверенности, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальные, согласованные, уникальные концы, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Клон без конца, без нити, уникальный, соответствие не установлено | |
| Клон без конца, плюс нить, уникальный, согласованный | |
| Клон без конца, без нити, множественный, соответствие не установлено | |
| Клон без конца, плюс нить, несогласованный, уникальный, не установлен |
4.Характеристики SNP
4.1 Цветовой код для дорожек SNP
В этой таблице описаны цвета по умолчанию для функций SNP. Эта окраска применяется к дорожкам dbSNP из базы данных NCBI SNP, а также к внешним / потоковым или загруженным файлам VCF.
Подробнее о рендеринге для треков ClinVar или GWAS см. Ниже.
| Тип модификации | Цвет |
|---|---|
| Однонуклеотидная вариация (SNV) | Красный |
| Удаление / вставка (DELINS) | Сине-фиолетовый |
| Удаление (DEL) | Синий |
| Вставка (INS) | желтый |
| Множественные вариации нуклеотидов (MNV) | Светло-серый |
4.1.1 Визуальные примеры
4.1.2 Вставка или удаление SNP
Дополнительное обозначение различает вставки и удаления.
Вставки отмечены двумя треугольниками в виде песочных часов:
Исключения отмечены треугольником, направленным вниз:
4.2 Визуализация треков ClinVar
дорожек данных VCF из проекта NCBI ClinVar имеют четкую окраску и визуализацию.
| Интерпретация ClinVar | Цвет |
|---|---|
| Патогенное | Темно-пурпурный |
| Вероятно патогенный | розовый |
| Доброкачественные | Темно-зеленый |
| Скорее всего доброкачественная | зеленый |
| Ответ на лекарства | Синий |
| Не предусмотрено; Неопределенное значение; Противоречивые интерпретации; Все остальные статусы ClinVar | Темно-серый |
Метки треков указывают на принадлежность вариации ClinVar и аллель ClinVar.
Дополнительную информацию, включая статус обзора ClinVar и прогнозируемые молекулярные последствия, можно найти во всплывающих подсказках к функциям.
4.3 Визуализация результатов общегеномной ассоциации
В этом разделе описывается отрисовка для анализа полногеномных ассоциаций (GWAS) из NCBI dbGaP.
Цвет представляет наивысшее значение p в этой ячейке.
| Диапазон значений p | Цвет |
|---|---|
| <2 | бирюзовый |
| 2–3 | Небесно-голубой |
| 3-4 | Синий |
| 4-5 | зеленый |
| 5-6 | желтый |
| 6-7 | Оранжевый |
| > 7 | Красный |
График разброса над ячейками отражает отдельные значения p.И цвет (который использует ту же цветовую схему), и расположение точки на оси Y соответствуют значению p.
5. Варианты конструкции
В этом разделе описывается отрисовка для структурных вариантов с идентификаторами NCBI dbVar.
5.1 Общий рендеринг
Существует четыре общих сценария для большинства вариантов (либо SV, либо SSV), как показано в таблице ниже. Однако также возможны смешанные случаи с определенной точкой останова на одном конце и неопределенным диапазоном точек останова на другом конце.Здесь мы используем (CNV SV) в качестве примера:
| Тип точки останова | Рендеринг | Визуальный пример |
|---|---|---|
| С разрешением точки останова | Полностью насыщенный цвет | |
| С определенным диапазоном точки останова | Прозрачный цвет для диапазонов контрольных точек | |
| С неопределенной точкой останова, но известной внешней границей | Треугольники, направленные друг к другу | |
| С неопределенной точкой останова, но известной внутренней границей | Треугольники, направленные друг от друга |
5.2 варианта типов вызовов (SSV) и типы регионов (SV)
| Тип | Комментарий | Визуальный пример |
|---|---|---|
| Изменение количества копий | Цвет: фиолетовый Четыре общих случая плюс CNV с длиной делеции (CNV) | |
| Увеличение числа копий или дублирование | Цвет: синий (усиление SSV) | |
| Утеря или удаление номера копии | Цвет: красный Последний вариант потери с длиной делеции (SSV потери) | |
| Вставка мобильного элемента или Вставка новой последовательности | Цвет: синий (вставка SV или SSV) | |
| Тандемное дублирование | Цвет: темно-коричневый (Eversion SV или SSV) | |
| инверсия | Цвет: светло-фиолетовый (инверсия SV или SSV) | |
| Перемещение | Цвет: светлый индиго с рисунком (Translocation SV или SSV) | |
| Комплекс | Цвет: светло-лазурный (Комплекс SSV) | |
| Вставка / удаление или отступление | Цвет: красный | |
| Неизвестно | Цвет: серый (Неизвестный SV или SSV) |
Примечание:
- Тип области SV «Вариант количества копий» может иметь дочерние элементы только для типов SSV «Увеличение количества копий» и / или «Потеря количества копий» — в любой комбинации.Если все дочерние SSV данного SV представляют собой увеличение количества копий или потерю количества копий, тогда SV будет окрашен так же, как окрашены дочерние элементы: синий для усиления и красный для потери, соответственно. Если дети представляют собой смесь этих двух типов, то SV будет окрашен в фиолетовый цвет.
СВ типа «Комплекс» может иметь:
детей всех ССВ типа «Комплекс», или
- дочерних элементов двух или более типов SSV в любой комбинации (за исключением «увеличения числа копий» и «потери числа копий», которые описаны выше)
5.3. Группа стилей визуализации для связанных структурных вариантов
5.3.1 Отрисовка по умолчанию с отображением родительского и дочернего элементов
5.3.2 Визуализация с опорными вариантами в упакованном виде
Если в поддерживающих вариантах имеется несколько типов, будет использоваться несколько цветов, чтобы отразить соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все поддерживаемые варианты.
5.3.3 Наложение всех поддерживающих вариантов на родительский вариант
Опорные варианты накладываются поверх родительского варианта, а самые короткие варианты — сверху. Цвета отражают соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все варианты.
5,4. Интерактивная визуализация дочерних вызовов в альтернативной области
Установить параметры рендеринга варианта Вкладка диалогового окна Настройка как «Показать родительский элемент, развернуть дочерние элементы по щелчку».
Sequence Viewer покажет знак плюса с количеством дочерних дорожек для каждой родительской дорожки.
Щелкните значок «плюс», и программа просмотра последовательности покажет все дочерние дорожки и отобразит знак минус вместо плюса на родительской дорожке.
Щелкните значок «минус», чтобы снова свернуть дочерние дорожки.
6. Сегментарное дублирование
| Атрибут идентификатора | Цвет | Пример |
|---|---|---|
| > 99.0 | Оранжевый | |
| > 98,0 | желтый | |
| > 90,0 | Серый | |
| <= 90,0 | Черный |
7. Выравнивания
Дорожки выравнивания содержат дискретные последовательности, попарно выровненные по отношению к сборке верхнего уровня. Эти треки получены из выравниваний последовательностей, представленных в BAM или аналогичных форматах.Выравнивание сборки-сборки и результаты NCBI BLAST также визуализируются в виде дорожек выравнивания в графическом представлении последовательности.
7.1 Дисплей выравнивания
Когда для параметра «Отображение выравнивания » установлено значение «Адаптивное» (по умолчанию), данные выравнивания представляются по-разному при разных уровнях масштабирования (рисунок 7.1.1). Отображение выравнивания также можно установить в режим «Упаковано» (всегда показывает покрытие или представление в виде скоплений), «Лестничная диаграмма» (одно выравнивание на строку) и «Показать все» (всегда показывает отдельные выравнивания).Эти параметры находятся в меню настроек отображения трека (Рисунок 7.3.1 и Таблица 7.3).
Рисунок 7.1.1 . Просмотр совмещений с различными уровнями масштабирования, когда Отображение выравнивания установлено на «Адаптивный». При более низких уровнях масштабирования выравнивания отображаются в виде графиков покрытия или наложения в масштабе 2 журнала. При более высоких уровнях масштабирования становятся видны отдельные выравнивания. Обратите внимание, что пробелы и несоответствия окрашены в красный цвет.
При высоких уровнях масштабирования становятся очевидными детали выравнивания.По умолчанию совпадающие базы и интроны окрашены в серый цвет, несоответствия и пробелы — в красный, а вставки представлены синими полосами (при более высоких уровнях масштабирования) или синими песочными часами с вертикальными синими полосами, пропорциональными количеству вставленных баз (рис. 7.1.2). Знаки «меньше» или «больше» (<или>) указывают ориентацию выравнивания относительно сборки.
Рисунок 7.1.2 . Легенда об особенностях центровки.
7.2 Подсказки по выравниванию
При наведении курсора на дискретное выравнивание появляется всплывающая подсказка, в которой приводится дополнительная информация о выравнивании (рисунок 7.2.1). Всплывающие подсказки сообщают о покрытии, идентичности и количестве несовпадений, интронов, невыровненных областей и пробелов в выравнивании. Покрытие рассчитывается как количество выровненных оснований, деленное на общую длину последовательности признаков, в то время как идентичность рассчитывается как количество совпадающих оснований, деленное на выровненную длину (исключая пробелы или интроны, без штрафа за пробелы).
Если возможно / применимо, также предоставляется строка CIGAR. Также есть ссылка на последовательность FASTA исходного выровненного объекта.Кроме того, невыровненные нуклеотиды 5 ‘и 3’ указаны в нижней части всплывающей подсказки, и их можно просмотреть, нажав кнопку «Просмотр».
Когда всплывающая подсказка создается рядом с положением вставки (вставок) в выравнивании, информация о вставке отображается в разделе «Невыровненные области» всплывающей подсказки. Все координаты сообщаются относительно выровненного объекта. В одной всплывающей подсказке сообщается до трех вставок.
Всплывающую подсказку можно «закрепить» с помощью канцелярской кнопки в верхнем левом углу.Вся информация, представленная во всплывающей подсказке, может быть выделена и скопирована в буфер обмена. Щелчок по лупе вверху сбрасывает графическое представление выбранного объекта.
Рисунок 7.2.1 . Всплывающая подсказка для функции выравнивания, сообщающей о покрытии, идентичности и количестве несоответствий, пропусков и невыровненных областей в выравнивании. Последовательность и координаты невыровненных регионов доступны в нижней части всплывающей подсказки. Информация для вставки (вставок) предоставляется, если всплывающая подсказка создается в позиции вставки.
7.3 Параметры отображения выравнивания
Доступ к настройкам отображения трека можно получить, щелкнув название трека или выбрав опцию Configure Tracks под кнопкой Tracks на панели инструментов. Меню настроек отображения треков (рисунок 7.3.1) для треков совмещения предоставляет множество различных вариантов отображения и анализа (таблица 7.3). Эти параметры могут включать в себя создание представления наложения, проецирование элементов (рисунок 7.3.5) и связывание пар сопряжений (рисунок 7.3.6). Когда выбрана опция Показать pileup , над трассой появляется представление pileup (рисунок 7.3.2). Раскрывающееся меню Pileup Display включает параметры для наложения, включая отображение графика совпадений / несоответствий (вид по умолчанию, рисунок 7.3.2), таблицы совпадений / несоответствий (рисунок 7.3.3) или таблицы ATGC (рисунок 7.3.2). 4). Выпадающее меню Unaligned Tails включает параметры для отображения / скрытия невыровненных длин хвоста и невыровненных последовательностей. Для треков, для которых доступны метаданные, пользователи также могут скрыть выравнивания (дубликаты, плохие чтения) (рисунок 7.3.7) или отсортируйте совпадения по цепям или гаплотипам.
Рисунок 7.3.1 . Настройки отображения треков для трасс выравнивания.
Дополнительные параметры отображения доступны для Выравнивание сборки и сборки дорожек, предоставленных NCBI. Эти дорожки состоят из выравниваний первого и второго прохода, которые по умолчанию будут отображаться на виде. Выравнивания с первым проходом представляют собой последовательности, которые имеют взаимное наилучшее выравнивание на обеих сборках (оценка взаимности 3).Выравнивания во втором проходе обеспечивают лучшее выравнивание на одной из сборок в другом месте (оценка взаимности 1 или 2). Раскрывающееся меню Сортировка выравниваний для дорожек выравнивания сборки-сборки содержит дополнительную опцию для сортировки вида дорожки по оценке взаимности (первый проход по сравнению со вторым проходом). Выравнивания второго прохода также можно скрыть из поля зрения, сняв флажок Показать выравнивания второго прохода (по умолчанию установлено). Оценка взаимности для дорожек выравнивания сборки-сборки также указывается во всплывающих подсказках отдельных выравниваний.
| Категория отображения | Параметры раскрывающегося меню |
|---|---|
| Отображение выравнивания | Adaptive Packed Лестница (одно выравнивание на ряд) Показать все |
| Pileup Display | График совпадения / несоответствия (количество) График совпадения / несоответствия (процент) График ATGC (количество) График ATGC (процент) Таблица совпадений / несоответствий (количество) Таблица совпадений / несоответствий (процент) Таблица ATGC (количество) Таблица ATGC (в процентах) |
| Метод оценки * | Оценка качества колонки ДНК Разница на основе частоты Показать различия Цвета нуклеиновых кислот Показатель качества Отключено |
| Невыровненные хвосты | Скрыть Показать длину хвоста Показать последовательность |
| Скрыть выравнивания * | Нет Дубликаты Плохие чтения Дубликаты / Плохие чтения |
| Сортировать выравнивания по * | Без сортировки Нить выравнивания Гаплотип Оценка взаимности |
Таблица 7.3 . Параметры отображения выравнивания находятся в раскрывающихся меню в меню настроек отображения трека. * Эти параметры могут быть неполными и могут различаться в зависимости от характера данных основного трека.
Рисунок 7.3.2 . Когда для параметра Pileup Display установлено значение «График совпадения / несоответствия» (по умолчанию), над выравниванием отображается график наложения. Красные отметки на графике обозначают несовпадения, а черные отметки обозначают пробелы. Отображение центровки на рисунке усечено.
Рисунок 7.3.3 . Когда для параметра Pileup Display установлено значение «Таблица соответствия / несоответствия (количество)», над выравниванием отображается таблица наложения. Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается счетчик интрона, пробела, несоответствия, совпадения и общего количества. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.4 . Когда Pileup Display установлен на «Таблица ATGC (счетчик)», таблица наложения отображается над выравниванием.Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается общее количество, A, T, G, C, пробел и интрон. Каждый нуклеотид представлен разным цветом; интенсивность цвета коррелирует с долей совпадений с этим нуклеотидом в этой позиции. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.5 . Функция выравнивания RefSeqGene (NG_029920.1) с выбранной опцией Project features показывает аннотированный транскрипт RefSeq (NM_), белок (NP_) и другие функции.
Рисунок 7.3.6 . Когда выбрана опция Link Mate Pairs , пары сопряжения последовательности, если это указано метаданными в дорожке, связаны красной пунктирной линией на дисплее выравнивания. Когда выбран параметр Показать метки , отображаются метки для пар сопряжений.
Рисунок 7.3.7 . Дубликаты ПЦР, если они указаны метаданными на дорожке, окрашиваются в фиолетовый цвет. Дубликаты ПЦР также могут быть указаны во всплывающей подсказке.Дублирующиеся трассы можно скрыть от просмотра, выбрав «Дубликаты» в раскрывающемся меню Скрыть трассы .
8. Дорожка последовательности
Дорожка последовательности представлена сплошной полосой при высоких уровнях масштабирования. Нажатие кнопки увеличения для перехода к последовательности (обозначено стрелкой ниже) изменяет уровень масштабирования для отображения последовательности.
Для нуклеотидных последовательностей показаны как первичная, так и комплементарная цепи. Метки 5 ’(обведены синим кружком) на каждом конце дорожки указывают ориентацию прядей.
По умолчанию дорожка последовательности расположена чуть ниже линейки, а метка дорожки скрыта. Положение дорожки можно изменить с помощью перетаскивания.
Настройка отображения дорожки для дорожки последовательности доступна на панели конфигурации дорожки и предоставляет возможность отображать метку для этой дорожки и цветовые промежутки по типу промежутка.
Если установлен флажок Цветовые промежутки по типу , промежутки будут окрашены в соответствии с аннотацией к соответствующему доступу INSDC для последовательности (т.е. аннотацию к записи плоского файла GenBank). Зазоры известной длины будут окрашены в фиолетовый цвет, зазоры неизвестной длины — в красный цвет, а загрязнения — в желтый.
8.1 Цветовой код сегмента
Для собранных последовательностей (например, геномных сборок) разные типы сегментов последовательности будут окрашены по-разному в зависимости от природы лежащего в основе компонента.
| Тип сегмента | Готовый HTGS | Осадка HTGS | WGS | Другое | Разрыв |
|---|---|---|---|---|---|
| Цвет | Синий | Оранжевый | зеленый | Серый | Черный |
Сегмент промежутка может быть другого цвета, кроме черного, если установлен флажок Цветовые промежутки по типу (см. Выше).
8.2 Ограничение Карта сайта
Карты распознавания рестрикционного фермента, например MAP_000012.1, можно рассматривать как треки последовательности с сайтами рестрикции, обозначенными зелеными треугольниками под полосой последовательности.
| Посмотреть | Визуальный пример |
|---|---|
| Увеличенное изображение | |
| Увеличенное изображение с всплывающей подсказкой (при наведении курсора мыши на сайт) |
9.Карта сегмента
Дорожки данных каркасов (контигов) и траекторий (компонентов) можно найти в группе вертикальных вкладок Sequence на панели конфигурации дорожек.
9.1 Подмости
9.2 Путь мозаики (компоненты)
Раскраска элементов траектории мозаики соответствует цветовой схеме из Раздела 8.1. Перекрывающиеся области, не входящие в контур, окрашены в желтый цвет.
10. Шесть кадровых трансляций
11.Размещение этикетки
Существует четыре глобальных варианта размещения меток: по умолчанию, боковая метка, верхняя метка и без метки. «По умолчанию» может означать разные настройки для разных объектов. Например, метка по умолчанию для трасс — это метка сверху, а настройка по умолчанию для элементов — это метка сбоку.
11.1 Боковая этикетка по сравнению с прядью
В режиме боковой надписи этикетка всегда размещается на стороне объекта размером 5 футов.
11.2 Примеры
12. Отрисовка гистограммы или графика
Гистограмма или графическое представление используется для визуализации данных, которые распределяются в виде количества данных на позицию.Эти данные часто представлены в формате Wig, bigWig или bedGraph. Типы данных, отображаемые в виде графиков, включают данные последовательности РНК, эпигеномные данные и данные о сохранении. Настройки отображения трека позволяют пользователям выбирать между стилями отображения («Гистограмма», «Тепловая карта», «Линейный график») и масштабом («Линейный», «Лог-база 10», «Лог-база e», «Лог-база 2») .
По умолчанию диапазон значений гистограммы или линейного графика соответствует локальным видимым минимальным и максимальным значениям на дисплее. Масштабирование, панорамирование или иное изменение отображаемой области приведет к соответствующей настройке минимального и максимального значений.
Пользователь может выбрать опцию Set Value Range для ручного управления диапазоном значений на дисплее. Этот параметр доступен, если для стиля отображения задано значение «Гистограмма» или «Линейный график», а для масштаба установлено значение «Линейный». Если поставщик данных установил минимальное и максимальное значения для трека, поля Min value и Max value будут предварительно заполнены этими значениями. Пользователь может применять предварительно заполненные диапазоны значений или настраивать диапазон значений вручную.Если трек является частью составной группы треков из концентратора треков, доступна опция массового применения настроек ко всем отображаемым трекам в составной группе.
После применения пользовательских минимальных и максимальных значений любые данные графика, выходящие за пределы установленного диапазона значений, будут обрезаны на дисплее и окрашены в красный цвет, чтобы предупредить пользователя. Когда параметр Set Value Range не выбран, диапазон значений вернется к использованию локального минимума и максимума на дисплее.
12.1 трек-концентраторы: multiWig Tracks
Некоторые концентраторы треков включают файлы multiWig, которые состоят из нескольких субтреков. Файлы MultiWig отображаются на графическом дисплее в виде объединенных графиков с каждым субтреком разным цветом (рисунок 12.1.1). Настройки отображения дорожек для дорожек multiWig предоставляют параметры для настройки масштаба и отображения субтреков в сложенном или объединенном формате.
При нажатии на легенду субтрека открывается диалоговое окно настроек отображения субтрека (рисунок 12.1.2). Пользователи могут настроить цвет, непрозрачность и стиль отображения отдельных фрагментов, которые перечислены в раскрывающемся меню. Также есть возможность скрыть отдельные субтреки.
Рисунок 12.1.1. Объединенные и составные представления файлов multiWig. Щелкните легенду, чтобы получить доступ к настройкам отображения субтрека.
Рисунок 12.1.2. Диалог настроек отображения субтрека. Выпадающее меню обеспечивает доступ к настройкам для всех субтреков в треке multiWig.
Легенда графического представления
Эта легенда относится к программе просмотра последовательности NCBI. NCBI Genome Workbench использует аналогичные графические представления.
- Визуализация общих функций
- Особенности модели гена
- Особенности размещения клонов
- Характеристики SNP
- Варианты конструкции
- Сегментарные дублирования
- Раскладки
- Дорожка последовательности
- Карта сегмента
- Шесть кадровых переводов
- Размещение этикетки
- Отрисовка гистограммы или графика
1.Отрисовка общих функций
В этом разделе описывается отрисовка, используемая для функций генов, моделей РНК и белков, регуляторных сайтов и большинства других типов функций. Эти функции часто происходят из файлов, отформатированных как BED, bigBED, GFF3, GTF, ASN.1 и аналогичные форматы.
Специализированный рендеринг для SNP, структурных вариантов и сегментных дубликатов описан в следующих разделах этой документации.
1.1 Код цвета элемента
Белые стрелки внутри объектов указывают направление объекта относительно последовательности верхнего уровня.Серые стрелки в интронах (например, в характеристиках РНК и кодирующей области) указывают направление сплайсинга.
1.2 Параметры отображения функций
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks .
Пользователи могут установить разрешение дисплея с помощью раскрывающегося меню Параметры рендеринга . По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
Параметр , связывающий параметры , обеспечивает различные виды отображения для дорожек, содержащих родительские и дочерние функции; т.е. «Показать все»; «Показать родителя, объединить потомков»; «Показывать детей, а не родителей»; «Показывать родителей, а не детей»; «Наложите детей на родителя». Изменение этих параметров может привести к отображению большего или меньшего количества функций на дорожке, в зависимости от базового источника данных на дорожке.
1.2.1 Отображение функции интрона
Дорожкиинтрона отображают сплайсинговые соединения экзон-экзон на основе анализа данных RNA-seq. Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Дорожки функцийIntron содержат дополнительные параметры отображения дорожек, в том числе возможность сортировать элементы стыка сращивания по количеству считываний сращивания или по направлению сращивания (прядям).Пользователи также могут упростить отображение этой дорожки, фильтруя данные по количеству совмещенных считываний.
1.3 Специальные стили визуализации
1.3.1 Псевдогеновые признаки
| Настройки дисплея | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Показать все | Полосы в зеленой полосе гена | |
| Без генной полосы | Зеленая полоса фона | |
| Показать на одной строке со структурой экзона, без генной полосы | Зеленая полоса фона |
1.3.2 Элементы с несовершенным соответствием сборке
| Пример | Визуальный эффект | Визуальные примеры |
|---|---|---|
| РНК или CDS с несоответствием последовательности относительно сборки | Затененный фон |
1.3.3 Элементы с частичным расположением аннотаций (например, из-за ошибок сборки или зазоров)
| Примеры | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Частичный 5 ‘конец | Черные стрелки («<<» или «>>») на конце 5 ‘ | |
| Частичный 3 ‘конец | Черные стрелки («<<" или ">>») на конце 3 ‘ | |
| Внутренние неточные концы | Черные стрелки («<<» и «>>») на внутренних соединениях | |
| Частичные 5- и 3-дюймовые концы | Черные стрелки («<<» и «>>») на обоих концах |
1.3.4 Элемент, помеченный как частичный
| Примеры случаев | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Элемент, отмеченный как частичный на концах 5 ’и 3’ (белые стрелки, «<<» и «>>»), и элемент с частичным концом 3 ’(черные стрелки« >> ») | Белые стрелки («<<» и «>>») на обоих концах или черные стрелки («>>») на одном конце. |
1.3.5 Ограничение возможностей сайта
Сайты узнавания рестрикционного фермента помечены треугольными метками, указывающими сайты разрезов.Треугольные метки отсутствуют при высоких уровнях масштабирования. Когда элемент выбирается щелчком мыши, расположение участков разреза обозначается красными линиями волос.
| Посмотреть | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Сайт узнавания рестрикционного фермента | Треугольники указывают места разрезов | |
| Место ограничения (функция выбрана) | Треугольники указывают место разреза; линиями волос отмечаются границы пространственных объектов и участки выреза | |
| Место ограничения (функция выбрана, уменьшено) | Без треугольников; линиями волос отмечаются границы пространственных объектов и участки выреза |
1.4 украшения
| Стиль декора | Визуальный эффект | Визуальные примеры |
|---|---|---|
| По умолчанию | Сплошные столбцы для интервалов признаков или экзонов и сплошные линии для интронов | |
| Стрелки | Стрелки на обоих концах, показывающие нить, и заштрихованные полосы для интронов | |
| Квадратный анкер | Квадрат для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Круглый анкер | Круг для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Необычный | Круг для начала только мРНК, квадрат для начала других признаков, кроме гена и CDS, стрелка для остановки функции, заштрихованные столбцы для интронов мРНК и кривые линии для интронов CDS |
2.Особенности модели гена
Характеристики генной модели состоят из нескольких возможных компонентов: генная панель, РНК / мРНК, CDS и характеристики экзона.
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks . Для треков аннотации генов NCBI или Ensembl могут быть доступны несколько вариантов. Параметр Hide non-coding скроет некодирующие транскрипты или некодирующие гены или псевдогены, чтобы отображались только варианты транскриптов, кодирующие белок. Hide model transcripts Функция скрывает модели стенограммы, предсказанные с помощью алгоритма Gnomon NCBI.
У вас также могут быть опции для отображения только вариантов, которые являются частью проекта CCDS или были обозначены как вариант RefSeq или MANE Select.
2.1 Визуализация генной модели
Настройки отображения трека включают несколько опций рендеринга, как описано ниже.
Когда все функции развернуты, столбцы генов окрашиваются в зеленый цвет, некодирующие РНК и элементы мРНК окрашиваются в фиолетовый цвет, а элементы CDS — в красный цвет.
При слиянии пар транскрипта и CDS кодирующие экзоны отображаются темно-зеленым цветом, а некодирующие области (т. Е. 5 ‘и 3’ UTR) отображаются более светлым оттенком зеленого, а некодирующие РНК остаются пурпурными (см. Пример «Объединить транскрипт и пары CDS, без генной полосы»).
Когда все транскрипты объединены и объединенные функции включают части экзонов, которые присутствуют в некоторых транскриптах, но отсутствуют в других транскриптах, интенсивность зеленой окраски пропорциональна количеству вариантов транскриптов, которые включают конкретную область экзона (см. «Объединить все транскрипты и CDS, без генной полосы », пример ниже).
Чтобы показать признаки, аннотированные на РНК или CDS, установите флажок Показать аннотированные признаки белка .
2.2 Специальная визуализация расхождений между аннотированными последовательностями RefSeq и геномными последовательностями
Визуализация несовпадений
В случае несинонимичных несовпадений, несоответствие выделяется комбинацией красного текста для кодона, отображения альтернативных оснований в мРНК и отображения альтернативных аминокислотных остатков в белковой характеристике.
В случае синонимичных несоответствий несоответствие выделяется комбинацией красного текста для кодона и отображения альтернативных оснований в мРНК.
Обработка вставок и удалений
Делеции отображаются в виде штрихов в соответствующих характеристиках мРНК и белков и отсутствуют какие-либо кодоны или аминокислоты.
Вставки отображаются в виде пары перевернутых вертикальных треугольников в мРНК и характеристиках белка, и показаны соответствующие кодоны и аминокислоты; кодоны окрашены в красный цвет.
2.3 Функциональная линейка
Когда все признаки развернуты в дорожке генной модели, при выборе объекта РНК или CDS, щелкнув по нему, появится линейка признаков, указывающая локальные координаты признака.
Когда выбрано несколько элементов, появляются линии волос, указывающие на различия в структуре разных элементов.
2.4 Подсказка по функциям
При наведении курсора на объект появляется всплывающая подсказка с дополнительной информацией об этом объекте.Всплывающая подсказка для функций генов, РНК и CDS сообщает о длине элемента, размахе сборки и координатах позиций, в которых всплывающая подсказка была активирована. Также включены ссылки на другие NCBI или внешние ресурсы, относящиеся к гену или функции, если таковые имеются. Также есть варианты для загрузки функции в форматах FASTA или GenBank, отправки ее в BLAST или открытия в ее собственном экземпляре графического средства просмотра последовательности. Более подробная информация о всплывающих подсказках для генов доступна здесь.
3. Особенности размещения клонов
3.1 Сообщаемые атрибуты
Графические изображения для всех элементов размещения клонов передают следующие атрибуты:
- Согласованность
- Уникальность
- Клонирование конца уверенности
- направленность и
- Подтверждающие доказательства
3.2 Визуальные примеры для переданного атрибута
3.3 Примеры визуализации для различных комбинаций атрибутов
Рендеринг может обрабатывать любую комбинацию пяти атрибутов, показанных выше.Ниже приведены несколько примеров рендеринга с различными комбинациями атрибутов.
| Дисплей | Описание |
|---|---|
| Уникальные, гармоничные, уникальные концы | |
| Множественный, согласованный, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, согласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, согласованный, один неуверенный конец, один виртуальный конец | |
| Уникальные, диссонирующие, уникальные концы | |
| Множественные, несогласованные, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, несогласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, противоречивый, один конец, не зависящий от уверенности, один виртуальный конец | |
| Уникальный, согласование не установлено, уникальные концы | |
| Множественные, согласование не установлено, один уникальный конец, один множественный конец | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец достоверности не установлена | |
| Уникальный, согласование не установлено, один конец с неопределенной достоверностью, один виртуальный конец | |
| Множественные, несогласованные, один уникальный конец, один множественный конец, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец с неопределенностью уверенности, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальные, согласованные, уникальные концы, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Клон без конца, без нити, уникальный, соответствие не установлено | |
| Клон без конца, плюс нить, уникальный, согласованный | |
| Клон без конца, без нити, множественный, соответствие не установлено | |
| Клон без конца, плюс нить, несогласованный, уникальный, не установлен |
4.Характеристики SNP
4.1 Цветовой код для дорожек SNP
В этой таблице описаны цвета по умолчанию для функций SNP. Эта окраска применяется к дорожкам dbSNP из базы данных NCBI SNP, а также к внешним / потоковым или загруженным файлам VCF.
Подробнее о рендеринге для треков ClinVar или GWAS см. Ниже.
| Тип модификации | Цвет |
|---|---|
| Однонуклеотидная вариация (SNV) | Красный |
| Удаление / вставка (DELINS) | Сине-фиолетовый |
| Удаление (DEL) | Синий |
| Вставка (INS) | желтый |
| Множественные вариации нуклеотидов (MNV) | Светло-серый |
4.1.1 Визуальные примеры
4.1.2 Вставка или удаление SNP
Дополнительное обозначение различает вставки и удаления.
Вставки отмечены двумя треугольниками в виде песочных часов:
Исключения отмечены треугольником, направленным вниз:
4.2 Визуализация треков ClinVar
дорожек данных VCF из проекта NCBI ClinVar имеют четкую окраску и визуализацию.
| Интерпретация ClinVar | Цвет |
|---|---|
| Патогенное | Темно-пурпурный |
| Вероятно патогенный | розовый |
| Доброкачественные | Темно-зеленый |
| Скорее всего доброкачественная | зеленый |
| Ответ на лекарства | Синий |
| Не предусмотрено; Неопределенное значение; Противоречивые интерпретации; Все остальные статусы ClinVar | Темно-серый |
Метки треков указывают на принадлежность вариации ClinVar и аллель ClinVar.
Дополнительную информацию, включая статус обзора ClinVar и прогнозируемые молекулярные последствия, можно найти во всплывающих подсказках к функциям.
4.3 Визуализация результатов общегеномной ассоциации
В этом разделе описывается отрисовка для анализа полногеномных ассоциаций (GWAS) из NCBI dbGaP.
Цвет представляет наивысшее значение p в этой ячейке.
| Диапазон значений p | Цвет |
|---|---|
| <2 | бирюзовый |
| 2–3 | Небесно-голубой |
| 3-4 | Синий |
| 4-5 | зеленый |
| 5-6 | желтый |
| 6-7 | Оранжевый |
| > 7 | Красный |
График разброса над ячейками отражает отдельные значения p.И цвет (который использует ту же цветовую схему), и расположение точки на оси Y соответствуют значению p.
5. Варианты конструкции
В этом разделе описывается отрисовка для структурных вариантов с идентификаторами NCBI dbVar.
5.1 Общий рендеринг
Существует четыре общих сценария для большинства вариантов (либо SV, либо SSV), как показано в таблице ниже. Однако также возможны смешанные случаи с определенной точкой останова на одном конце и неопределенным диапазоном точек останова на другом конце.Здесь мы используем (CNV SV) в качестве примера:
| Тип точки останова | Рендеринг | Визуальный пример |
|---|---|---|
| С разрешением точки останова | Полностью насыщенный цвет | |
| С определенным диапазоном точки останова | Прозрачный цвет для диапазонов контрольных точек | |
| С неопределенной точкой останова, но известной внешней границей | Треугольники, направленные друг к другу | |
| С неопределенной точкой останова, но известной внутренней границей | Треугольники, направленные друг от друга |
5.2 варианта типов вызовов (SSV) и типы регионов (SV)
| Тип | Комментарий | Визуальный пример |
|---|---|---|
| Изменение количества копий | Цвет: фиолетовый Четыре общих случая плюс CNV с длиной делеции (CNV) | |
| Увеличение числа копий или дублирование | Цвет: синий (усиление SSV) | |
| Утеря или удаление номера копии | Цвет: красный Последний вариант потери с длиной делеции (SSV потери) | |
| Вставка мобильного элемента или Вставка новой последовательности | Цвет: синий (вставка SV или SSV) | |
| Тандемное дублирование | Цвет: темно-коричневый (Eversion SV или SSV) | |
| инверсия | Цвет: светло-фиолетовый (инверсия SV или SSV) | |
| Перемещение | Цвет: светлый индиго с рисунком (Translocation SV или SSV) | |
| Комплекс | Цвет: светло-лазурный (Комплекс SSV) | |
| Вставка / удаление или отступление | Цвет: красный | |
| Неизвестно | Цвет: серый (Неизвестный SV или SSV) |
Примечание:
- Тип области SV «Вариант количества копий» может иметь дочерние элементы только для типов SSV «Увеличение количества копий» и / или «Потеря количества копий» — в любой комбинации.Если все дочерние SSV данного SV представляют собой увеличение количества копий или потерю количества копий, тогда SV будет окрашен так же, как окрашены дочерние элементы: синий для усиления и красный для потери, соответственно. Если дети представляют собой смесь этих двух типов, то SV будет окрашен в фиолетовый цвет.
СВ типа «Комплекс» может иметь:
детей всех ССВ типа «Комплекс», или
- дочерних элементов двух или более типов SSV в любой комбинации (за исключением «увеличения числа копий» и «потери числа копий», которые описаны выше)
5.3. Группа стилей визуализации для связанных структурных вариантов
5.3.1 Отрисовка по умолчанию с отображением родительского и дочернего элементов
5.3.2 Визуализация с опорными вариантами в упакованном виде
Если в поддерживающих вариантах имеется несколько типов, будет использоваться несколько цветов, чтобы отразить соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все поддерживаемые варианты.
5.3.3 Наложение всех поддерживающих вариантов на родительский вариант
Опорные варианты накладываются поверх родительского варианта, а самые короткие варианты — сверху. Цвета отражают соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все варианты.
5,4. Интерактивная визуализация дочерних вызовов в альтернативной области
Установить параметры рендеринга варианта Вкладка диалогового окна Настройка как «Показать родительский элемент, развернуть дочерние элементы по щелчку».
Sequence Viewer покажет знак плюса с количеством дочерних дорожек для каждой родительской дорожки.
Щелкните значок «плюс», и программа просмотра последовательности покажет все дочерние дорожки и отобразит знак минус вместо плюса на родительской дорожке.
Щелкните значок «минус», чтобы снова свернуть дочерние дорожки.
6. Сегментарное дублирование
| Атрибут идентификатора | Цвет | Пример |
|---|---|---|
| > 99.0 | Оранжевый | |
| > 98,0 | желтый | |
| > 90,0 | Серый | |
| <= 90,0 | Черный |
7. Выравнивания
Дорожки выравнивания содержат дискретные последовательности, попарно выровненные по отношению к сборке верхнего уровня. Эти треки получены из выравниваний последовательностей, представленных в BAM или аналогичных форматах.Выравнивание сборки-сборки и результаты NCBI BLAST также визуализируются в виде дорожек выравнивания в графическом представлении последовательности.
7.1 Дисплей выравнивания
Когда для параметра «Отображение выравнивания » установлено значение «Адаптивное» (по умолчанию), данные выравнивания представляются по-разному при разных уровнях масштабирования (рисунок 7.1.1). Отображение выравнивания также можно установить в режим «Упаковано» (всегда показывает покрытие или представление в виде скоплений), «Лестничная диаграмма» (одно выравнивание на строку) и «Показать все» (всегда показывает отдельные выравнивания).Эти параметры находятся в меню настроек отображения трека (Рисунок 7.3.1 и Таблица 7.3).
Рисунок 7.1.1 . Просмотр совмещений с различными уровнями масштабирования, когда Отображение выравнивания установлено на «Адаптивный». При более низких уровнях масштабирования выравнивания отображаются в виде графиков покрытия или наложения в масштабе 2 журнала. При более высоких уровнях масштабирования становятся видны отдельные выравнивания. Обратите внимание, что пробелы и несоответствия окрашены в красный цвет.
При высоких уровнях масштабирования становятся очевидными детали выравнивания.По умолчанию совпадающие базы и интроны окрашены в серый цвет, несоответствия и пробелы — в красный, а вставки представлены синими полосами (при более высоких уровнях масштабирования) или синими песочными часами с вертикальными синими полосами, пропорциональными количеству вставленных баз (рис. 7.1.2). Знаки «меньше» или «больше» (<или>) указывают ориентацию выравнивания относительно сборки.
Рисунок 7.1.2 . Легенда об особенностях центровки.
7.2 Подсказки по выравниванию
При наведении курсора на дискретное выравнивание появляется всплывающая подсказка, в которой приводится дополнительная информация о выравнивании (рисунок 7.2.1). Всплывающие подсказки сообщают о покрытии, идентичности и количестве несовпадений, интронов, невыровненных областей и пробелов в выравнивании. Покрытие рассчитывается как количество выровненных оснований, деленное на общую длину последовательности признаков, в то время как идентичность рассчитывается как количество совпадающих оснований, деленное на выровненную длину (исключая пробелы или интроны, без штрафа за пробелы).
Если возможно / применимо, также предоставляется строка CIGAR. Также есть ссылка на последовательность FASTA исходного выровненного объекта.Кроме того, невыровненные нуклеотиды 5 ‘и 3’ указаны в нижней части всплывающей подсказки, и их можно просмотреть, нажав кнопку «Просмотр».
Когда всплывающая подсказка создается рядом с положением вставки (вставок) в выравнивании, информация о вставке отображается в разделе «Невыровненные области» всплывающей подсказки. Все координаты сообщаются относительно выровненного объекта. В одной всплывающей подсказке сообщается до трех вставок.
Всплывающую подсказку можно «закрепить» с помощью канцелярской кнопки в верхнем левом углу.Вся информация, представленная во всплывающей подсказке, может быть выделена и скопирована в буфер обмена. Щелчок по лупе вверху сбрасывает графическое представление выбранного объекта.
Рисунок 7.2.1 . Всплывающая подсказка для функции выравнивания, сообщающей о покрытии, идентичности и количестве несоответствий, пропусков и невыровненных областей в выравнивании. Последовательность и координаты невыровненных регионов доступны в нижней части всплывающей подсказки. Информация для вставки (вставок) предоставляется, если всплывающая подсказка создается в позиции вставки.
7.3 Параметры отображения выравнивания
Доступ к настройкам отображения трека можно получить, щелкнув название трека или выбрав опцию Configure Tracks под кнопкой Tracks на панели инструментов. Меню настроек отображения треков (рисунок 7.3.1) для треков совмещения предоставляет множество различных вариантов отображения и анализа (таблица 7.3). Эти параметры могут включать в себя создание представления наложения, проецирование элементов (рисунок 7.3.5) и связывание пар сопряжений (рисунок 7.3.6). Когда выбрана опция Показать pileup , над трассой появляется представление pileup (рисунок 7.3.2). Раскрывающееся меню Pileup Display включает параметры для наложения, включая отображение графика совпадений / несоответствий (вид по умолчанию, рисунок 7.3.2), таблицы совпадений / несоответствий (рисунок 7.3.3) или таблицы ATGC (рисунок 7.3.2). 4). Выпадающее меню Unaligned Tails включает параметры для отображения / скрытия невыровненных длин хвоста и невыровненных последовательностей. Для треков, для которых доступны метаданные, пользователи также могут скрыть выравнивания (дубликаты, плохие чтения) (рисунок 7.3.7) или отсортируйте совпадения по цепям или гаплотипам.
Рисунок 7.3.1 . Настройки отображения треков для трасс выравнивания.
Дополнительные параметры отображения доступны для Выравнивание сборки и сборки дорожек, предоставленных NCBI. Эти дорожки состоят из выравниваний первого и второго прохода, которые по умолчанию будут отображаться на виде. Выравнивания с первым проходом представляют собой последовательности, которые имеют взаимное наилучшее выравнивание на обеих сборках (оценка взаимности 3).Выравнивания во втором проходе обеспечивают лучшее выравнивание на одной из сборок в другом месте (оценка взаимности 1 или 2). Раскрывающееся меню Сортировка выравниваний для дорожек выравнивания сборки-сборки содержит дополнительную опцию для сортировки вида дорожки по оценке взаимности (первый проход по сравнению со вторым проходом). Выравнивания второго прохода также можно скрыть из поля зрения, сняв флажок Показать выравнивания второго прохода (по умолчанию установлено). Оценка взаимности для дорожек выравнивания сборки-сборки также указывается во всплывающих подсказках отдельных выравниваний.
| Категория отображения | Параметры раскрывающегося меню |
|---|---|
| Отображение выравнивания | Adaptive Packed Лестница (одно выравнивание на ряд) Показать все |
| Pileup Display | График совпадения / несоответствия (количество) График совпадения / несоответствия (процент) График ATGC (количество) График ATGC (процент) Таблица совпадений / несоответствий (количество) Таблица совпадений / несоответствий (процент) Таблица ATGC (количество) Таблица ATGC (в процентах) |
| Метод оценки * | Оценка качества колонки ДНК Разница на основе частоты Показать различия Цвета нуклеиновых кислот Показатель качества Отключено |
| Невыровненные хвосты | Скрыть Показать длину хвоста Показать последовательность |
| Скрыть выравнивания * | Нет Дубликаты Плохие чтения Дубликаты / Плохие чтения |
| Сортировать выравнивания по * | Без сортировки Нить выравнивания Гаплотип Оценка взаимности |
Таблица 7.3 . Параметры отображения выравнивания находятся в раскрывающихся меню в меню настроек отображения трека. * Эти параметры могут быть неполными и могут различаться в зависимости от характера данных основного трека.
Рисунок 7.3.2 . Когда для параметра Pileup Display установлено значение «График совпадения / несоответствия» (по умолчанию), над выравниванием отображается график наложения. Красные отметки на графике обозначают несовпадения, а черные отметки обозначают пробелы. Отображение центровки на рисунке усечено.
Рисунок 7.3.3 . Когда для параметра Pileup Display установлено значение «Таблица соответствия / несоответствия (количество)», над выравниванием отображается таблица наложения. Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается счетчик интрона, пробела, несоответствия, совпадения и общего количества. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.4 . Когда Pileup Display установлен на «Таблица ATGC (счетчик)», таблица наложения отображается над выравниванием.Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается общее количество, A, T, G, C, пробел и интрон. Каждый нуклеотид представлен разным цветом; интенсивность цвета коррелирует с долей совпадений с этим нуклеотидом в этой позиции. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.5 . Функция выравнивания RefSeqGene (NG_029920.1) с выбранной опцией Project features показывает аннотированный транскрипт RefSeq (NM_), белок (NP_) и другие функции.
Рисунок 7.3.6 . Когда выбрана опция Link Mate Pairs , пары сопряжения последовательности, если это указано метаданными в дорожке, связаны красной пунктирной линией на дисплее выравнивания. Когда выбран параметр Показать метки , отображаются метки для пар сопряжений.
Рисунок 7.3.7 . Дубликаты ПЦР, если они указаны метаданными на дорожке, окрашиваются в фиолетовый цвет. Дубликаты ПЦР также могут быть указаны во всплывающей подсказке.Дублирующиеся трассы можно скрыть от просмотра, выбрав «Дубликаты» в раскрывающемся меню Скрыть трассы .
8. Дорожка последовательности
Дорожка последовательности представлена сплошной полосой при высоких уровнях масштабирования. Нажатие кнопки увеличения для перехода к последовательности (обозначено стрелкой ниже) изменяет уровень масштабирования для отображения последовательности.
Для нуклеотидных последовательностей показаны как первичная, так и комплементарная цепи. Метки 5 ’(обведены синим кружком) на каждом конце дорожки указывают ориентацию прядей.
По умолчанию дорожка последовательности расположена чуть ниже линейки, а метка дорожки скрыта. Положение дорожки можно изменить с помощью перетаскивания.
Настройка отображения дорожки для дорожки последовательности доступна на панели конфигурации дорожки и предоставляет возможность отображать метку для этой дорожки и цветовые промежутки по типу промежутка.
Если установлен флажок Цветовые промежутки по типу , промежутки будут окрашены в соответствии с аннотацией к соответствующему доступу INSDC для последовательности (т.е. аннотацию к записи плоского файла GenBank). Зазоры известной длины будут окрашены в фиолетовый цвет, зазоры неизвестной длины — в красный цвет, а загрязнения — в желтый.
8.1 Цветовой код сегмента
Для собранных последовательностей (например, геномных сборок) разные типы сегментов последовательности будут окрашены по-разному в зависимости от природы лежащего в основе компонента.
| Тип сегмента | Готовый HTGS | Осадка HTGS | WGS | Другое | Разрыв |
|---|---|---|---|---|---|
| Цвет | Синий | Оранжевый | зеленый | Серый | Черный |
Сегмент промежутка может быть другого цвета, кроме черного, если установлен флажок Цветовые промежутки по типу (см. Выше).
8.2 Ограничение Карта сайта
Карты распознавания рестрикционного фермента, например MAP_000012.1, можно рассматривать как треки последовательности с сайтами рестрикции, обозначенными зелеными треугольниками под полосой последовательности.
| Посмотреть | Визуальный пример |
|---|---|
| Увеличенное изображение | |
| Увеличенное изображение с всплывающей подсказкой (при наведении курсора мыши на сайт) |
9.Карта сегмента
Дорожки данных каркасов (контигов) и траекторий (компонентов) можно найти в группе вертикальных вкладок Sequence на панели конфигурации дорожек.
9.1 Подмости
9.2 Путь мозаики (компоненты)
Раскраска элементов траектории мозаики соответствует цветовой схеме из Раздела 8.1. Перекрывающиеся области, не входящие в контур, окрашены в желтый цвет.
10. Шесть кадровых трансляций
11.Размещение этикетки
Существует четыре глобальных варианта размещения меток: по умолчанию, боковая метка, верхняя метка и без метки. «По умолчанию» может означать разные настройки для разных объектов. Например, метка по умолчанию для трасс — это метка сверху, а настройка по умолчанию для элементов — это метка сбоку.
11.1 Боковая этикетка по сравнению с прядью
В режиме боковой надписи этикетка всегда размещается на стороне объекта размером 5 футов.
11.2 Примеры
12. Отрисовка гистограммы или графика
Гистограмма или графическое представление используется для визуализации данных, которые распределяются в виде количества данных на позицию.Эти данные часто представлены в формате Wig, bigWig или bedGraph. Типы данных, отображаемые в виде графиков, включают данные последовательности РНК, эпигеномные данные и данные о сохранении. Настройки отображения трека позволяют пользователям выбирать между стилями отображения («Гистограмма», «Тепловая карта», «Линейный график») и масштабом («Линейный», «Лог-база 10», «Лог-база e», «Лог-база 2») .
По умолчанию диапазон значений гистограммы или линейного графика соответствует локальным видимым минимальным и максимальным значениям на дисплее. Масштабирование, панорамирование или иное изменение отображаемой области приведет к соответствующей настройке минимального и максимального значений.
Пользователь может выбрать опцию Set Value Range для ручного управления диапазоном значений на дисплее. Этот параметр доступен, если для стиля отображения задано значение «Гистограмма» или «Линейный график», а для масштаба установлено значение «Линейный». Если поставщик данных установил минимальное и максимальное значения для трека, поля Min value и Max value будут предварительно заполнены этими значениями. Пользователь может применять предварительно заполненные диапазоны значений или настраивать диапазон значений вручную.Если трек является частью составной группы треков из концентратора треков, доступна опция массового применения настроек ко всем отображаемым трекам в составной группе.
После применения пользовательских минимальных и максимальных значений любые данные графика, выходящие за пределы установленного диапазона значений, будут обрезаны на дисплее и окрашены в красный цвет, чтобы предупредить пользователя. Когда параметр Set Value Range не выбран, диапазон значений вернется к использованию локального минимума и максимума на дисплее.
12.1 трек-концентраторы: multiWig Tracks
Некоторые концентраторы треков включают файлы multiWig, которые состоят из нескольких субтреков. Файлы MultiWig отображаются на графическом дисплее в виде объединенных графиков с каждым субтреком разным цветом (рисунок 12.1.1). Настройки отображения дорожек для дорожек multiWig предоставляют параметры для настройки масштаба и отображения субтреков в сложенном или объединенном формате.
При нажатии на легенду субтрека открывается диалоговое окно настроек отображения субтрека (рисунок 12.1.2). Пользователи могут настроить цвет, непрозрачность и стиль отображения отдельных фрагментов, которые перечислены в раскрывающемся меню. Также есть возможность скрыть отдельные субтреки.
Рисунок 12.1.1. Объединенные и составные представления файлов multiWig. Щелкните легенду, чтобы получить доступ к настройкам отображения субтрека.
Рисунок 12.1.2. Диалог настроек отображения субтрека. Выпадающее меню обеспечивает доступ к настройкам для всех субтреков в треке multiWig.
Легенда графического представления
Эта легенда относится к программе просмотра последовательности NCBI. NCBI Genome Workbench использует аналогичные графические представления.
- Визуализация общих функций
- Особенности модели гена
- Особенности размещения клонов
- Характеристики SNP
- Варианты конструкции
- Сегментарные дублирования
- Раскладки
- Дорожка последовательности
- Карта сегмента
- Шесть кадровых переводов
- Размещение этикетки
- Отрисовка гистограммы или графика
1.Отрисовка общих функций
В этом разделе описывается отрисовка, используемая для функций генов, моделей РНК и белков, регуляторных сайтов и большинства других типов функций. Эти функции часто происходят из файлов, отформатированных как BED, bigBED, GFF3, GTF, ASN.1 и аналогичные форматы.
Специализированный рендеринг для SNP, структурных вариантов и сегментных дубликатов описан в следующих разделах этой документации.
1.1 Код цвета элемента
Белые стрелки внутри объектов указывают направление объекта относительно последовательности верхнего уровня.Серые стрелки в интронах (например, в характеристиках РНК и кодирующей области) указывают направление сплайсинга.
1.2 Параметры отображения функций
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks .
Пользователи могут установить разрешение дисплея с помощью раскрывающегося меню Параметры рендеринга . По умолчанию для большинства дорожек функций установлено значение «Упаковать функции при необходимости», при этом отдельные функции отображаются при увеличении масштаба, а данные отображаются в виде гистограммы при уменьшении.Параметр «Все объекты в одной строке» всегда отображает данные в виде гистограммы, а параметр «Все объекты расширены» всегда отображает дискретные объекты.
Параметр , связывающий параметры , обеспечивает различные виды отображения для дорожек, содержащих родительские и дочерние функции; т.е. «Показать все»; «Показать родителя, объединить потомков»; «Показывать детей, а не родителей»; «Показывать родителей, а не детей»; «Наложите детей на родителя». Изменение этих параметров может привести к отображению большего или меньшего количества функций на дорожке, в зависимости от базового источника данных на дорожке.
1.2.1 Отображение функции интрона
Дорожкиинтрона отображают сплайсинговые соединения экзон-экзон на основе анализа данных RNA-seq. Концы элементов соответствуют стыкам сращивания, а стрелка соответствует направлению сращивания. Количество считываний RNA-seq, поддерживающих соединение сплайсинга, сообщается для каждой функции. Наведите указатель мыши на объект, чтобы увидеть всплывающую подсказку с дополнительной информацией, включая конечные координаты и длину интрона.
Дорожки функцийIntron содержат дополнительные параметры отображения дорожек, в том числе возможность сортировать элементы стыка сращивания по количеству считываний сращивания или по направлению сращивания (прядям).Пользователи также могут упростить отображение этой дорожки, фильтруя данные по количеству совмещенных считываний.
1.3 Специальные стили визуализации
1.3.1 Псевдогеновые признаки
| Настройки дисплея | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Показать все | Полосы в зеленой полосе гена | |
| Без генной полосы | Зеленая полоса фона | |
| Показать на одной строке со структурой экзона, без генной полосы | Зеленая полоса фона |
1.3.2 Элементы с несовершенным соответствием сборке
| Пример | Визуальный эффект | Визуальные примеры |
|---|---|---|
| РНК или CDS с несоответствием последовательности относительно сборки | Затененный фон |
1.3.3 Элементы с частичным расположением аннотаций (например, из-за ошибок сборки или зазоров)
| Примеры | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Частичный 5 ‘конец | Черные стрелки («<<» или «>>») на конце 5 ‘ | |
| Частичный 3 ‘конец | Черные стрелки («<<" или ">>») на конце 3 ‘ | |
| Внутренние неточные концы | Черные стрелки («<<» и «>>») на внутренних соединениях | |
| Частичные 5- и 3-дюймовые концы | Черные стрелки («<<» и «>>») на обоих концах |
1.3.4 Элемент, помеченный как частичный
| Примеры случаев | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Элемент, отмеченный как частичный на концах 5 ’и 3’ (белые стрелки, «<<» и «>>»), и элемент с частичным концом 3 ’(черные стрелки« >> ») | Белые стрелки («<<» и «>>») на обоих концах или черные стрелки («>>») на одном конце. |
1.3.5 Ограничение возможностей сайта
Сайты узнавания рестрикционного фермента помечены треугольными метками, указывающими сайты разрезов.Треугольные метки отсутствуют при высоких уровнях масштабирования. Когда элемент выбирается щелчком мыши, расположение участков разреза обозначается красными линиями волос.
| Посмотреть | Визуальный эффект | Визуальные примеры |
|---|---|---|
| Сайт узнавания рестрикционного фермента | Треугольники указывают места разрезов | |
| Место ограничения (функция выбрана) | Треугольники указывают место разреза; линиями волос отмечаются границы пространственных объектов и участки выреза | |
| Место ограничения (функция выбрана, уменьшено) | Без треугольников; линиями волос отмечаются границы пространственных объектов и участки выреза |
1.4 украшения
| Стиль декора | Визуальный эффект | Визуальные примеры |
|---|---|---|
| По умолчанию | Сплошные столбцы для интервалов признаков или экзонов и сплошные линии для интронов | |
| Стрелки | Стрелки на обоих концах, показывающие нить, и заштрихованные полосы для интронов | |
| Квадратный анкер | Квадрат для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Круглый анкер | Круг для начала функции, стрелка для остановки функции, пунктирные линии для интронов | |
| Необычный | Круг для начала только мРНК, квадрат для начала других признаков, кроме гена и CDS, стрелка для остановки функции, заштрихованные столбцы для интронов мРНК и кривые линии для интронов CDS |
2.Особенности модели гена
Характеристики генной модели состоят из нескольких возможных компонентов: генная панель, РНК / мРНК, CDS и характеристики экзона.
К диалоговому окну настроек отображения трека можно получить доступ, щелкнув название трека, значок шестеренки справа от трека или выбрав трек в меню конфигурации Tracks . Для треков аннотации генов NCBI или Ensembl могут быть доступны несколько вариантов. Параметр Hide non-coding скроет некодирующие транскрипты или некодирующие гены или псевдогены, чтобы отображались только варианты транскриптов, кодирующие белок. Hide model transcripts Функция скрывает модели стенограммы, предсказанные с помощью алгоритма Gnomon NCBI.
У вас также могут быть опции для отображения только вариантов, которые являются частью проекта CCDS или были обозначены как вариант RefSeq или MANE Select.
2.1 Визуализация генной модели
Настройки отображения трека включают несколько опций рендеринга, как описано ниже.
Когда все функции развернуты, столбцы генов окрашиваются в зеленый цвет, некодирующие РНК и элементы мРНК окрашиваются в фиолетовый цвет, а элементы CDS — в красный цвет.
При слиянии пар транскрипта и CDS кодирующие экзоны отображаются темно-зеленым цветом, а некодирующие области (т. Е. 5 ‘и 3’ UTR) отображаются более светлым оттенком зеленого, а некодирующие РНК остаются пурпурными (см. Пример «Объединить транскрипт и пары CDS, без генной полосы»).
Когда все транскрипты объединены и объединенные функции включают части экзонов, которые присутствуют в некоторых транскриптах, но отсутствуют в других транскриптах, интенсивность зеленой окраски пропорциональна количеству вариантов транскриптов, которые включают конкретную область экзона (см. «Объединить все транскрипты и CDS, без генной полосы », пример ниже).
Чтобы показать признаки, аннотированные на РНК или CDS, установите флажок Показать аннотированные признаки белка .
2.2 Специальная визуализация расхождений между аннотированными последовательностями RefSeq и геномными последовательностями
Визуализация несовпадений
В случае несинонимичных несовпадений, несоответствие выделяется комбинацией красного текста для кодона, отображения альтернативных оснований в мРНК и отображения альтернативных аминокислотных остатков в белковой характеристике.
В случае синонимичных несоответствий несоответствие выделяется комбинацией красного текста для кодона и отображения альтернативных оснований в мРНК.
Обработка вставок и удалений
Делеции отображаются в виде штрихов в соответствующих характеристиках мРНК и белков и отсутствуют какие-либо кодоны или аминокислоты.
Вставки отображаются в виде пары перевернутых вертикальных треугольников в мРНК и характеристиках белка, и показаны соответствующие кодоны и аминокислоты; кодоны окрашены в красный цвет.
2.3 Функциональная линейка
Когда все признаки развернуты в дорожке генной модели, при выборе объекта РНК или CDS, щелкнув по нему, появится линейка признаков, указывающая локальные координаты признака.
Когда выбрано несколько элементов, появляются линии волос, указывающие на различия в структуре разных элементов.
2.4 Подсказка по функциям
При наведении курсора на объект появляется всплывающая подсказка с дополнительной информацией об этом объекте.Всплывающая подсказка для функций генов, РНК и CDS сообщает о длине элемента, размахе сборки и координатах позиций, в которых всплывающая подсказка была активирована. Также включены ссылки на другие NCBI или внешние ресурсы, относящиеся к гену или функции, если таковые имеются. Также есть варианты для загрузки функции в форматах FASTA или GenBank, отправки ее в BLAST или открытия в ее собственном экземпляре графического средства просмотра последовательности. Более подробная информация о всплывающих подсказках для генов доступна здесь.
3. Особенности размещения клонов
3.1 Сообщаемые атрибуты
Графические изображения для всех элементов размещения клонов передают следующие атрибуты:
- Согласованность
- Уникальность
- Клонирование конца уверенности
- направленность и
- Подтверждающие доказательства
3.2 Визуальные примеры для переданного атрибута
3.3 Примеры визуализации для различных комбинаций атрибутов
Рендеринг может обрабатывать любую комбинацию пяти атрибутов, показанных выше.Ниже приведены несколько примеров рендеринга с различными комбинациями атрибутов.
| Дисплей | Описание |
|---|---|
| Уникальные, гармоничные, уникальные концы | |
| Множественный, согласованный, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, согласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, согласованный, один неуверенный конец, один виртуальный конец | |
| Уникальные, диссонирующие, уникальные концы | |
| Множественные, несогласованные, один уникальный конец, один множественный конец | |
| Неустановленная уникальность, несогласованная, один множественный конец, один конец достоверности неустановленной | |
| Уникальный, противоречивый, один конец, не зависящий от уверенности, один виртуальный конец | |
| Уникальный, согласование не установлено, уникальные концы | |
| Множественные, согласование не установлено, один уникальный конец, один множественный конец | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец достоверности не установлена | |
| Уникальный, согласование не установлено, один конец с неопределенной достоверностью, один виртуальный конец | |
| Множественные, несогласованные, один уникальный конец, один множественный конец, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальность не установлена, согласованность не установлена, один множественный конец, один конец с неопределенностью уверенности, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Уникальные, согласованные, уникальные концы, не все концы, не являющиеся прототипами, являются «поддерживающими» | |
| Клон без конца, без нити, уникальный, соответствие не установлено | |
| Клон без конца, плюс нить, уникальный, согласованный | |
| Клон без конца, без нити, множественный, соответствие не установлено | |
| Клон без конца, плюс нить, несогласованный, уникальный, не установлен |
4.Характеристики SNP
4.1 Цветовой код для дорожек SNP
В этой таблице описаны цвета по умолчанию для функций SNP. Эта окраска применяется к дорожкам dbSNP из базы данных NCBI SNP, а также к внешним / потоковым или загруженным файлам VCF.
Подробнее о рендеринге для треков ClinVar или GWAS см. Ниже.
| Тип модификации | Цвет |
|---|---|
| Однонуклеотидная вариация (SNV) | Красный |
| Удаление / вставка (DELINS) | Сине-фиолетовый |
| Удаление (DEL) | Синий |
| Вставка (INS) | желтый |
| Множественные вариации нуклеотидов (MNV) | Светло-серый |
4.1.1 Визуальные примеры
4.1.2 Вставка или удаление SNP
Дополнительное обозначение различает вставки и удаления.
Вставки отмечены двумя треугольниками в виде песочных часов:
Исключения отмечены треугольником, направленным вниз:
4.2 Визуализация треков ClinVar
дорожек данных VCF из проекта NCBI ClinVar имеют четкую окраску и визуализацию.
| Интерпретация ClinVar | Цвет |
|---|---|
| Патогенное | Темно-пурпурный |
| Вероятно патогенный | розовый |
| Доброкачественные | Темно-зеленый |
| Скорее всего доброкачественная | зеленый |
| Ответ на лекарства | Синий |
| Не предусмотрено; Неопределенное значение; Противоречивые интерпретации; Все остальные статусы ClinVar | Темно-серый |
Метки треков указывают на принадлежность вариации ClinVar и аллель ClinVar.
Дополнительную информацию, включая статус обзора ClinVar и прогнозируемые молекулярные последствия, можно найти во всплывающих подсказках к функциям.
4.3 Визуализация результатов общегеномной ассоциации
В этом разделе описывается отрисовка для анализа полногеномных ассоциаций (GWAS) из NCBI dbGaP.
Цвет представляет наивысшее значение p в этой ячейке.
| Диапазон значений p | Цвет |
|---|---|
| <2 | бирюзовый |
| 2–3 | Небесно-голубой |
| 3-4 | Синий |
| 4-5 | зеленый |
| 5-6 | желтый |
| 6-7 | Оранжевый |
| > 7 | Красный |
График разброса над ячейками отражает отдельные значения p.И цвет (который использует ту же цветовую схему), и расположение точки на оси Y соответствуют значению p.
5. Варианты конструкции
В этом разделе описывается отрисовка для структурных вариантов с идентификаторами NCBI dbVar.
5.1 Общий рендеринг
Существует четыре общих сценария для большинства вариантов (либо SV, либо SSV), как показано в таблице ниже. Однако также возможны смешанные случаи с определенной точкой останова на одном конце и неопределенным диапазоном точек останова на другом конце.Здесь мы используем (CNV SV) в качестве примера:
| Тип точки останова | Рендеринг | Визуальный пример |
|---|---|---|
| С разрешением точки останова | Полностью насыщенный цвет | |
| С определенным диапазоном точки останова | Прозрачный цвет для диапазонов контрольных точек | |
| С неопределенной точкой останова, но известной внешней границей | Треугольники, направленные друг к другу | |
| С неопределенной точкой останова, но известной внутренней границей | Треугольники, направленные друг от друга |
5.2 варианта типов вызовов (SSV) и типы регионов (SV)
| Тип | Комментарий | Визуальный пример |
|---|---|---|
| Изменение количества копий | Цвет: фиолетовый Четыре общих случая плюс CNV с длиной делеции (CNV) | |
| Увеличение числа копий или дублирование | Цвет: синий (усиление SSV) | |
| Утеря или удаление номера копии | Цвет: красный Последний вариант потери с длиной делеции (SSV потери) | |
| Вставка мобильного элемента или Вставка новой последовательности | Цвет: синий (вставка SV или SSV) | |
| Тандемное дублирование | Цвет: темно-коричневый (Eversion SV или SSV) | |
| инверсия | Цвет: светло-фиолетовый (инверсия SV или SSV) | |
| Перемещение | Цвет: светлый индиго с рисунком (Translocation SV или SSV) | |
| Комплекс | Цвет: светло-лазурный (Комплекс SSV) | |
| Вставка / удаление или отступление | Цвет: красный | |
| Неизвестно | Цвет: серый (Неизвестный SV или SSV) |
Примечание:
- Тип области SV «Вариант количества копий» может иметь дочерние элементы только для типов SSV «Увеличение количества копий» и / или «Потеря количества копий» — в любой комбинации.Если все дочерние SSV данного SV представляют собой увеличение количества копий или потерю количества копий, тогда SV будет окрашен так же, как окрашены дочерние элементы: синий для усиления и красный для потери, соответственно. Если дети представляют собой смесь этих двух типов, то SV будет окрашен в фиолетовый цвет.
СВ типа «Комплекс» может иметь:
детей всех ССВ типа «Комплекс», или
- дочерних элементов двух или более типов SSV в любой комбинации (за исключением «увеличения числа копий» и «потери числа копий», которые описаны выше)
5.3. Группа стилей визуализации для связанных структурных вариантов
5.3.1 Отрисовка по умолчанию с отображением родительского и дочернего элементов
5.3.2 Визуализация с опорными вариантами в упакованном виде
Если в поддерживающих вариантах имеется несколько типов, будет использоваться несколько цветов, чтобы отразить соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все поддерживаемые варианты.
5.3.3 Наложение всех поддерживающих вариантов на родительский вариант
Опорные варианты накладываются поверх родительского варианта, а самые короткие варианты — сверху. Цвета отражают соответствующий тип аллеля.
Щелкните и выберите упакованную панель функций, чтобы отобразить все варианты.
5,4. Интерактивная визуализация дочерних вызовов в альтернативной области
Установить параметры рендеринга варианта Вкладка диалогового окна Настройка как «Показать родительский элемент, развернуть дочерние элементы по щелчку».
Sequence Viewer покажет знак плюса с количеством дочерних дорожек для каждой родительской дорожки.
Щелкните значок «плюс», и программа просмотра последовательности покажет все дочерние дорожки и отобразит знак минус вместо плюса на родительской дорожке.
Щелкните значок «минус», чтобы снова свернуть дочерние дорожки.
6. Сегментарное дублирование
| Атрибут идентификатора | Цвет | Пример |
|---|---|---|
| > 99.0 | Оранжевый | |
| > 98,0 | желтый | |
| > 90,0 | Серый | |
| <= 90,0 | Черный |
7. Выравнивания
Дорожки выравнивания содержат дискретные последовательности, попарно выровненные по отношению к сборке верхнего уровня. Эти треки получены из выравниваний последовательностей, представленных в BAM или аналогичных форматах.Выравнивание сборки-сборки и результаты NCBI BLAST также визуализируются в виде дорожек выравнивания в графическом представлении последовательности.
7.1 Дисплей выравнивания
Когда для параметра «Отображение выравнивания » установлено значение «Адаптивное» (по умолчанию), данные выравнивания представляются по-разному при разных уровнях масштабирования (рисунок 7.1.1). Отображение выравнивания также можно установить в режим «Упаковано» (всегда показывает покрытие или представление в виде скоплений), «Лестничная диаграмма» (одно выравнивание на строку) и «Показать все» (всегда показывает отдельные выравнивания).Эти параметры находятся в меню настроек отображения трека (Рисунок 7.3.1 и Таблица 7.3).
Рисунок 7.1.1 . Просмотр совмещений с различными уровнями масштабирования, когда Отображение выравнивания установлено на «Адаптивный». При более низких уровнях масштабирования выравнивания отображаются в виде графиков покрытия или наложения в масштабе 2 журнала. При более высоких уровнях масштабирования становятся видны отдельные выравнивания. Обратите внимание, что пробелы и несоответствия окрашены в красный цвет.
При высоких уровнях масштабирования становятся очевидными детали выравнивания.По умолчанию совпадающие базы и интроны окрашены в серый цвет, несоответствия и пробелы — в красный, а вставки представлены синими полосами (при более высоких уровнях масштабирования) или синими песочными часами с вертикальными синими полосами, пропорциональными количеству вставленных баз (рис. 7.1.2). Знаки «меньше» или «больше» (<или>) указывают ориентацию выравнивания относительно сборки.
Рисунок 7.1.2 . Легенда об особенностях центровки.
7.2 Подсказки по выравниванию
При наведении курсора на дискретное выравнивание появляется всплывающая подсказка, в которой приводится дополнительная информация о выравнивании (рисунок 7.2.1). Всплывающие подсказки сообщают о покрытии, идентичности и количестве несовпадений, интронов, невыровненных областей и пробелов в выравнивании. Покрытие рассчитывается как количество выровненных оснований, деленное на общую длину последовательности признаков, в то время как идентичность рассчитывается как количество совпадающих оснований, деленное на выровненную длину (исключая пробелы или интроны, без штрафа за пробелы).
Если возможно / применимо, также предоставляется строка CIGAR. Также есть ссылка на последовательность FASTA исходного выровненного объекта.Кроме того, невыровненные нуклеотиды 5 ‘и 3’ указаны в нижней части всплывающей подсказки, и их можно просмотреть, нажав кнопку «Просмотр».
Когда всплывающая подсказка создается рядом с положением вставки (вставок) в выравнивании, информация о вставке отображается в разделе «Невыровненные области» всплывающей подсказки. Все координаты сообщаются относительно выровненного объекта. В одной всплывающей подсказке сообщается до трех вставок.
Всплывающую подсказку можно «закрепить» с помощью канцелярской кнопки в верхнем левом углу.Вся информация, представленная во всплывающей подсказке, может быть выделена и скопирована в буфер обмена. Щелчок по лупе вверху сбрасывает графическое представление выбранного объекта.
Рисунок 7.2.1 . Всплывающая подсказка для функции выравнивания, сообщающей о покрытии, идентичности и количестве несоответствий, пропусков и невыровненных областей в выравнивании. Последовательность и координаты невыровненных регионов доступны в нижней части всплывающей подсказки. Информация для вставки (вставок) предоставляется, если всплывающая подсказка создается в позиции вставки.
7.3 Параметры отображения выравнивания
Доступ к настройкам отображения трека можно получить, щелкнув название трека или выбрав опцию Configure Tracks под кнопкой Tracks на панели инструментов. Меню настроек отображения треков (рисунок 7.3.1) для треков совмещения предоставляет множество различных вариантов отображения и анализа (таблица 7.3). Эти параметры могут включать в себя создание представления наложения, проецирование элементов (рисунок 7.3.5) и связывание пар сопряжений (рисунок 7.3.6). Когда выбрана опция Показать pileup , над трассой появляется представление pileup (рисунок 7.3.2). Раскрывающееся меню Pileup Display включает параметры для наложения, включая отображение графика совпадений / несоответствий (вид по умолчанию, рисунок 7.3.2), таблицы совпадений / несоответствий (рисунок 7.3.3) или таблицы ATGC (рисунок 7.3.2). 4). Выпадающее меню Unaligned Tails включает параметры для отображения / скрытия невыровненных длин хвоста и невыровненных последовательностей. Для треков, для которых доступны метаданные, пользователи также могут скрыть выравнивания (дубликаты, плохие чтения) (рисунок 7.3.7) или отсортируйте совпадения по цепям или гаплотипам.
Рисунок 7.3.1 . Настройки отображения треков для трасс выравнивания.
Дополнительные параметры отображения доступны для Выравнивание сборки и сборки дорожек, предоставленных NCBI. Эти дорожки состоят из выравниваний первого и второго прохода, которые по умолчанию будут отображаться на виде. Выравнивания с первым проходом представляют собой последовательности, которые имеют взаимное наилучшее выравнивание на обеих сборках (оценка взаимности 3).Выравнивания во втором проходе обеспечивают лучшее выравнивание на одной из сборок в другом месте (оценка взаимности 1 или 2). Раскрывающееся меню Сортировка выравниваний для дорожек выравнивания сборки-сборки содержит дополнительную опцию для сортировки вида дорожки по оценке взаимности (первый проход по сравнению со вторым проходом). Выравнивания второго прохода также можно скрыть из поля зрения, сняв флажок Показать выравнивания второго прохода (по умолчанию установлено). Оценка взаимности для дорожек выравнивания сборки-сборки также указывается во всплывающих подсказках отдельных выравниваний.
| Категория отображения | Параметры раскрывающегося меню |
|---|---|
| Отображение выравнивания | Adaptive Packed Лестница (одно выравнивание на ряд) Показать все |
| Pileup Display | График совпадения / несоответствия (количество) График совпадения / несоответствия (процент) График ATGC (количество) График ATGC (процент) Таблица совпадений / несоответствий (количество) Таблица совпадений / несоответствий (процент) Таблица ATGC (количество) Таблица ATGC (в процентах) |
| Метод оценки * | Оценка качества колонки ДНК Разница на основе частоты Показать различия Цвета нуклеиновых кислот Показатель качества Отключено |
| Невыровненные хвосты | Скрыть Показать длину хвоста Показать последовательность |
| Скрыть выравнивания * | Нет Дубликаты Плохие чтения Дубликаты / Плохие чтения |
| Сортировать выравнивания по * | Без сортировки Нить выравнивания Гаплотип Оценка взаимности |
Таблица 7.3 . Параметры отображения выравнивания находятся в раскрывающихся меню в меню настроек отображения трека. * Эти параметры могут быть неполными и могут различаться в зависимости от характера данных основного трека.
Рисунок 7.3.2 . Когда для параметра Pileup Display установлено значение «График совпадения / несоответствия» (по умолчанию), над выравниванием отображается график наложения. Красные отметки на графике обозначают несовпадения, а черные отметки обозначают пробелы. Отображение центровки на рисунке усечено.
Рисунок 7.3.3 . Когда для параметра Pileup Display установлено значение «Таблица соответствия / несоответствия (количество)», над выравниванием отображается таблица наложения. Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается счетчик интрона, пробела, несоответствия, совпадения и общего количества. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.4 . Когда Pileup Display установлен на «Таблица ATGC (счетчик)», таблица наложения отображается над выравниванием.Когда масштаб установлен на уровень последовательности, для каждой позиции сообщается общее количество, A, T, G, C, пробел и интрон. Каждый нуклеотид представлен разным цветом; интенсивность цвета коррелирует с долей совпадений с этим нуклеотидом в этой позиции. Пустая запись означает, что счетчик равен 0. Отображение выравнивания на рисунке усечено.
Рисунок 7.3.5 . Функция выравнивания RefSeqGene (NG_029920.1) с выбранной опцией Project features показывает аннотированный транскрипт RefSeq (NM_), белок (NP_) и другие функции.
Рисунок 7.3.6 . Когда выбрана опция Link Mate Pairs , пары сопряжения последовательности, если это указано метаданными в дорожке, связаны красной пунктирной линией на дисплее выравнивания. Когда выбран параметр Показать метки , отображаются метки для пар сопряжений.
Рисунок 7.3.7 . Дубликаты ПЦР, если они указаны метаданными на дорожке, окрашиваются в фиолетовый цвет. Дубликаты ПЦР также могут быть указаны во всплывающей подсказке.Дублирующиеся трассы можно скрыть от просмотра, выбрав «Дубликаты» в раскрывающемся меню Скрыть трассы .
8. Дорожка последовательности
Дорожка последовательности представлена сплошной полосой при высоких уровнях масштабирования. Нажатие кнопки увеличения для перехода к последовательности (обозначено стрелкой ниже) изменяет уровень масштабирования для отображения последовательности.
Для нуклеотидных последовательностей показаны как первичная, так и комплементарная цепи. Метки 5 ’(обведены синим кружком) на каждом конце дорожки указывают ориентацию прядей.
По умолчанию дорожка последовательности расположена чуть ниже линейки, а метка дорожки скрыта. Положение дорожки можно изменить с помощью перетаскивания.
Настройка отображения дорожки для дорожки последовательности доступна на панели конфигурации дорожки и предоставляет возможность отображать метку для этой дорожки и цветовые промежутки по типу промежутка.
Если установлен флажок Цветовые промежутки по типу , промежутки будут окрашены в соответствии с аннотацией к соответствующему доступу INSDC для последовательности (т.е. аннотацию к записи плоского файла GenBank). Зазоры известной длины будут окрашены в фиолетовый цвет, зазоры неизвестной длины — в красный цвет, а загрязнения — в желтый.
8.1 Цветовой код сегмента
Для собранных последовательностей (например, геномных сборок) разные типы сегментов последовательности будут окрашены по-разному в зависимости от природы лежащего в основе компонента.
| Тип сегмента | Готовый HTGS | Осадка HTGS | WGS | Другое | Разрыв |
|---|---|---|---|---|---|
| Цвет | Синий | Оранжевый | зеленый | Серый | Черный |
Сегмент промежутка может быть другого цвета, кроме черного, если установлен флажок Цветовые промежутки по типу (см. Выше).
8.2 Ограничение Карта сайта
Карты распознавания рестрикционного фермента, например MAP_000012.1, можно рассматривать как треки последовательности с сайтами рестрикции, обозначенными зелеными треугольниками под полосой последовательности.
| Посмотреть | Визуальный пример |
|---|---|
| Увеличенное изображение | |
| Увеличенное изображение с всплывающей подсказкой (при наведении курсора мыши на сайт) |
9.Карта сегмента
Дорожки данных каркасов (контигов) и траекторий (компонентов) можно найти в группе вертикальных вкладок Sequence на панели конфигурации дорожек.
9.1 Подмости
9.2 Путь мозаики (компоненты)
Раскраска элементов траектории мозаики соответствует цветовой схеме из Раздела 8.1. Перекрывающиеся области, не входящие в контур, окрашены в желтый цвет.
10. Шесть кадровых трансляций
11.Размещение этикетки
Существует четыре глобальных варианта размещения меток: по умолчанию, боковая метка, верхняя метка и без метки. «По умолчанию» может означать разные настройки для разных объектов. Например, метка по умолчанию для трасс — это метка сверху, а настройка по умолчанию для элементов — это метка сбоку.
11.1 Боковая этикетка по сравнению с прядью
В режиме боковой надписи этикетка всегда размещается на стороне объекта размером 5 футов.
11.2 Примеры
12. Отрисовка гистограммы или графика
Гистограмма или графическое представление используется для визуализации данных, которые распределяются в виде количества данных на позицию.Эти данные часто представлены в формате Wig, bigWig или bedGraph. Типы данных, отображаемые в виде графиков, включают данные последовательности РНК, эпигеномные данные и данные о сохранении. Настройки отображения трека позволяют пользователям выбирать между стилями отображения («Гистограмма», «Тепловая карта», «Линейный график») и масштабом («Линейный», «Лог-база 10», «Лог-база e», «Лог-база 2») .
По умолчанию диапазон значений гистограммы или линейного графика соответствует локальным видимым минимальным и максимальным значениям на дисплее. Масштабирование, панорамирование или иное изменение отображаемой области приведет к соответствующей настройке минимального и максимального значений.
Пользователь может выбрать опцию Set Value Range для ручного управления диапазоном значений на дисплее. Этот параметр доступен, если для стиля отображения задано значение «Гистограмма» или «Линейный график», а для масштаба установлено значение «Линейный». Если поставщик данных установил минимальное и максимальное значения для трека, поля Min value и Max value будут предварительно заполнены этими значениями. Пользователь может применять предварительно заполненные диапазоны значений или настраивать диапазон значений вручную.Если трек является частью составной группы треков из концентратора треков, доступна опция массового применения настроек ко всем отображаемым трекам в составной группе.
После применения пользовательских минимальных и максимальных значений любые данные графика, выходящие за пределы установленного диапазона значений, будут обрезаны на дисплее и окрашены в красный цвет, чтобы предупредить пользователя. Когда параметр Set Value Range не выбран, диапазон значений вернется к использованию локального минимума и максимума на дисплее.
12.1 трек-концентраторы: multiWig Tracks
Некоторые концентраторы треков включают файлы multiWig, которые состоят из нескольких субтреков. Файлы MultiWig отображаются на графическом дисплее в виде объединенных графиков с каждым субтреком разным цветом (рисунок 12.1.1). Настройки отображения дорожек для дорожек multiWig предоставляют параметры для настройки масштаба и отображения субтреков в сложенном или объединенном формате.
При нажатии на легенду субтрека открывается диалоговое окно настроек отображения субтрека (рисунок 12.1.2). Пользователи могут настроить цвет, непрозрачность и стиль отображения отдельных фрагментов, которые перечислены в раскрывающемся меню. Также есть возможность скрыть отдельные субтреки.
Рисунок 12.1.1. Объединенные и составные представления файлов multiWig. Щелкните легенду, чтобы получить доступ к настройкам отображения субтрека.
Рисунок 12.1.2. Диалог настроек отображения субтрека. Выпадающее меню обеспечивает доступ к настройкам для всех субтреков в треке multiWig.
Глава 17. Инструменты графического интерфейса пользователя для управления гостевыми виртуальными машинами Red Hat Enterprise Linux 6
virt-viewer — это минималистичная утилита командной строки для отображения графической консоли гостевой виртуальной машины. Доступ к консоли осуществляется по протоколу VNC или SPICE. На гостя можно ссылаться по его имени, идентификатору или UUID. Если гость еще не запущен, средство просмотра можно настроить на ожидание запуска, прежде чем пытаться подключиться к консоли.Средство просмотра может подключаться к удаленным хостам для получения информации о консоли, а затем также подключаться к удаленной консоли, используя тот же сетевой транспорт.
По сравнению с virt-manager , virt-viewer предлагает меньший набор функций, но менее требователен к ресурсам. Кроме того, в отличие от virt-manager , virt-viewer в большинстве случаев не требует разрешений на чтение и запись для libvirt. Следовательно, его могут использовать непривилегированные пользователи, которые должны иметь возможность подключаться и отображать гостей, но не для их настройки.
Чтобы установить утилиту virt-viewer, запустите:
# sudo yum install virt-viewer
Синтаксис
Базовый синтаксис командной строки virt-viewer выглядит следующим образом:
# virt-viewer [ОПЦИИ] {guest-name | id | uuid} Базовый синтаксис командной строки virt-viewer выглядит следующим образом:
Подключение к гостевой виртуальной машине
При использовании без каких-либо опций virt-viewer выводит список гостей, к которым он может подключиться на гипервизоре по умолчанию в локальной системе.
Чтобы подключиться к гостевой виртуальной машине, которая использует гипервизор по умолчанию:# virt-viewer имя-гостя-или-UUIDЧтобы подключиться к гостевой виртуальной машине, которая использует гипервизор KVM-QEMU:
# virt-viewer --connect qemu: /// system имя-гостя-или-UUIDЧтобы подключиться к удаленной консоли с помощью TLS:
# virt-viewer --connect xen: //example.org/ имя-гостя-или-UUIDЧтобы подключиться к консоли на удаленном хосте с помощью SSH, найдите гостевую конфигурацию, а затем выполните прямое соединение с консолью без туннелирования:
# virt-viewer --direct --connect xen + ssh: // root @ example.org / имя-гостя или UUID
Интерфейс
По умолчанию интерфейс virt-viewer предоставляет только базовые инструменты для взаимодействия с гостем:
Рисунок 17.1. Пример virt-viewer interface
Настройка горячих клавиш
Чтобы создать настраиваемое сочетание клавиш (также называемое горячей клавишей) для сеанса virt-viewer , используйте параметр --hotkeys :
# virt-viewer --hotkeys = action1 = комбинация клавиш1 [, action2 = комбинация клавиш2 ] имя гостя или UUID
Горячей клавише можно назначить следующие действия:
Включить полноэкранный режим
релиз-курсор
смарт-карта-вставка
смарт-карта-удалить
Горячие клавиши комбинации клавиш не чувствительны к регистру.Обратите внимание, что настройка горячих клавиш не переносится на будущие сеансы virt-viewer .
Пример 17.1. Настройка virt-viewer hotkey
Чтобы добавить горячую клавишу для перехода в полноэкранный режим при подключении к гостевой системе KVM-QEMU с именем testguest:
# virt-viewer --hotkeys = toggle-fullscreen = shift + f11 qemu: /// system testguest
Режим киоска
В режиме киоска virt-viewer позволяет пользователю взаимодействовать только с подключенным рабочим столом и не предоставляет никаких параметров для взаимодействия с гостевыми настройками или хост-системой, если гость не выключен.Это может быть полезно, например, когда администратор хочет ограничить диапазон действий пользователя указанным гостем.
Чтобы использовать режим киоска, подключитесь к гостю с опцией -k или --kiosk .
Пример 17.2. Использование virt-viewer в режиме киоска
Чтобы подключиться к виртуальной машине KVM-QEMU в режиме киоска, который завершается после выключения машины, используйте следующую команду:
# virt-viewer --connect qemu: /// system guest-name-or-UUID --kiosk --kiosk-quit on-disconnect
Обратите внимание, однако, что режим киоска сам по себе не может гарантировать, что пользователь не взаимодействует с хост-системой или гостевыми настройками после выключения гостя.Это потребует дополнительных мер безопасности, таких как отключение оконного менеджера на хосте.
soft -atter / pimsviewer: графический интерфейс пользователя (GUI) для PIMS
Графический интерфейс пользователя (GUI) для PIMS (снимок экрана ниже)
Эта программа просмотра основана на PyQt5 и может работать с N-мерными файлами изображений, открытыми PIMS.
Кроме того, он имеет инфраструктуру плагинов, которая может использоваться для расширения основных функций.
Установка
Pimsviewer можно установить с помощью conda:
conda install -c conda-forge pimsviewer
В качестве альтернативы его также можно установить с помощью pip:
При установке последней исходной версии всегда устанавливайте ее с pip (а не с python setup.py develop , это приведет к ошибкам зависимости для PyQt ):
Обычная установка:
Установочная установка:
Запуск программы просмотра
После установки программы просмотра доступен исполняемый файл pimsviewer . Просто запустите команду через интерфейс терминала / командной строки.
$ pimsviewer - справка
Использование: pimsviewer [ОПЦИИ] [FILEPATH]
Параметры:
--example-plugins / --no-example-plugins
Загрузите дополнительные примеры плагинов
--help Показать это сообщение и выйти. Скриншот
Примеры
Все приведенные ниже примеры также доступны в виде файлов сценариев в папке examples .
Запустив pimsviewer --example-plugins , вы можете предварительно просмотреть примеры подключаемых модулей, используемых ниже.
Пример 00: Использование средства просмотра из Python
Вы можете использовать программу просмотра в скрипте Python следующим образом:
импорт систем
из графического интерфейса импорта pimsviewer
из PyQt5.QtWidgets импортировать QApplication
filepath = 'путь / к / файлу'
# Имена классов дополнительных плагинов для добавления
плагины = []
app = QApplication (sys.argv)
gui = GUI (extra_plugins = плагины)
gui.open (имя_файла = путь к файлу)
gui.show ()
sys.exit (app.exec_ ())
Пример 01: Использование средства просмотра из Python (более короткий путь)
Или, если вам не нужна ссылка на реальный объект, но вы просто хотите запустить программу:
из pimsviewer import run
запустить ('путь / к / файлу')
В обоих случаях вы можете не указывать путь к файлу.
Пример 02: оценка эффекта функции обработки
В этом примере добавлена функция обработки, которая добавляет регулируемое количество шума. к изображению.Уровень шума регулируется ползунком.
из pimsviewer import run
из pimsviewer.plugins import ProcessingPlugin
run ('путь / к / файлу', [ProcessingPlugin])
Пример 03: аннотирование объектов на видео
В этом примере аннотируются функции, полученные с помощью trackpy, на видео. Отслеживаемые позиции загружаются пользователем из файла CSV pandas DataFrame.
из pimsviewer import run
из pimsviewer.plugins import AnnotatePlugin
run ('путь / к / файлу', [AnnotatePlugin])
Ваш собственный плагин?
Посмотрев на код примеров плагинов, должно быть довольно легко расширьте pimsviewer, используя свои собственные плагины.Свяжитесь с одним из сопровождающих, если вы есть проблемы с написанием собственных плагинов.
Pimsviewer версии 1.0 написал Каспер ван дер Вел, версии, начиная с 2.0, написал Рубен Вервей.
.
 д.), выделена одна из функций, то в предварительном просмотре графики будет отображена та страница, на которой находится эта функция. Позиция такой выделяется.
д.), выделена одна из функций, то в предварительном просмотре графики будет отображена та страница, на которой находится эта функция. Позиция такой выделяется.

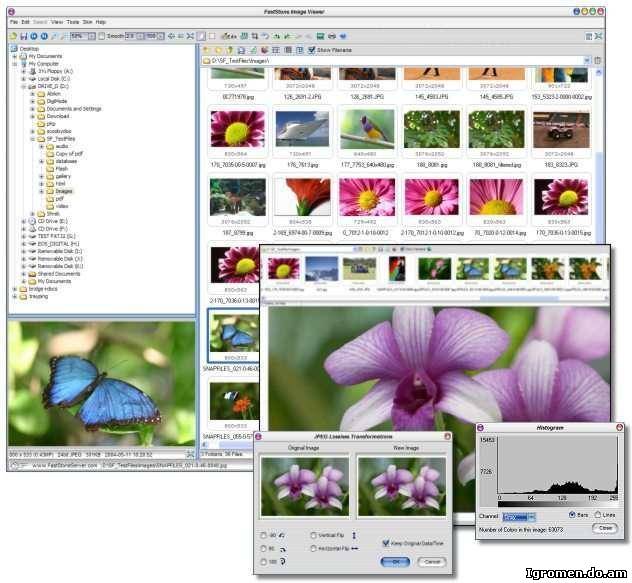







Ваш комментарий будет первым